Categoria bioinformática
Kit de laboratório doméstico de engenharia genética
Descrição do Produto
NOTA: A maneira como podemos oferecer este kit a um preço tão baixo é encontrando as melhores ofertas com nossos parceiros e fabricantes. Isso significa que este kit leva de 1 a 2 meses para ser enviado. Por favor, seja paciente e entenda que estamos fazendo o possível para chegar até você.
Consulte-nos sobre códigos de desconto para nossas aulas on-line com a compra deste kit.
Escolha nossa opção de plano de pagamento no check-out para fazer 4 pagamentos mensais.Veja mais informações aqui .
Este kit inicial do laboratório de bricolage fornece todo o equipamento, reagentes e materiais necessários para começar em biologia molecular e engenharia genética. Além disso, inclui um kit de genotipagem e suprimentos do nosso kit DIY CRISPR para que você possa realizar seus primeiros experimentos! Vem com tutoriais explicando a ciência e como usar o equipamento.
Este kit vem com
- Máquina de PCR com tampa aquecida
- Tubos de PCR
- Um conjunto de 3 novas pipetas de grau laboratorial
- Pontas de pipeta para cada pipeta
- Caixa de eletroforese em gel de agarose e molde de gel com pente
- Rack de tubo
- Uma fonte de alimentação para executar a eletroforese
- 2 powerchords
- Almofada de aquecimento com temperatura controlada para culturas em crescimento
- 10g Agarose
- 50g de mistura de tampão TAE (27g de base Tris / 23g de acetato de Tris / 0,5g de EDTA)
- 5g de sulfato de canamicina
- 5g de ampicilina de sódio
- 40g de ágar LB
- 20g LB Media
- Manga de 20 placas de Petri
- Balanças de 0,01g – 200g e balanças
- Óculos trans-eye-luminator e luz azul
- 5 x 15mL Tubes
- 5 x tubos de 50 ml
- bactérias ativadas por luz pDusk, pDawn
- pJE202 bactérias brilhantes bioluminescentes geneticamente modificadas
- 100uL Gel Verde Mancha 10.000x
- 500uL 5x Taq Master Mix
- Corante de carregamento de DNA de 200 uL
- 100uL 100bp DNA Ladder
- 100uL 1kbp DNA Ladder
- Kit de Genotipagem
- Suprimentos do kit DIY CRISPR
- Microcentrífuga 10k novíssima
Modelagem por homologia de receptores canabinóides CB1 e CB2 e estudos de docking
AUTORES: da Silva, J.A. (UNIVERSIDADE FEDERAL DE ALAGOAS) ; Silva, T.M.B. () ; Balliano, T.L. ()
RESUMO:Avaliou se os dados obtidos com os disponíveis na literatura, de moléculas como potenciais inibidores para receptores canabinóides CB1 e CB2, onde foi feito um estudo de modelagem molecular com CB1 e CB2, cuja estrutura tridimensional não disponível em banco de dados, foi obtida por modelagem por homologia; seguido de uma simulação de docking para o qual usamos moléculas de estrutura do sistema monoterpeno
PALAVRAS CHAVES: Modelagem por homologia; receptores canabinóides; docking
INTRODUÇÃO:No planejamento de um fármaco tem como base a informação estrutural do bioreceptor que permite a descoberta e síntese de compostos com complementaridade estérica, hidrofóbica e eletrostática ao seu sítio de ligação. Em todo este processo usamos a modelagem molecular como ferramenta. Como para os receptores canabinóides CB1 e CB2, o alvo protéico macromolecular ainda não foi determinado experimentalmente, construímos os modelos em um procedimento comparativo conhecido como modelagem molecular por homologia estrutural (SANTOS-FILHO; DEALENCASTRO,2003). Os canabinóides são uma classe de compostos que exerçam efeitos farmacológicos diretos sobre um determinado número de órgãos e normalmente relacionados ao sistema nervoso central.
MATERIAL E MÉTODOS:Para a obtenção dos modelos das proteínas CB1 e CB2 foi usado arquivos FASTA obtidos do banco de dados NCBI, então foi usado o software Modeller (SALI; BLUNDELL, 1993). Em seguida foi feito a simulação de docking como programa Autodock 4.2 (que usa algoritmo de busca, o algoritmo genético Lamarkiano), com um conjunto de moléculas das quais os melhores scores foi obtido com o ligante 1: 1- Hidroxi-6,6-dimetil-3-pentil-6H-benzo[c]cromeno-9-ácido carboxílico e ligante 2: 6,6,9-Trimetil-3-pentil-6H –benzo[c]cromeno-1,8-diol. O ancoramento foi verificado no sítio de ligação previsto por LIGSITE (HENDLICH; RIPPMANN, 1997). Desta forma obtemos dados de energia de ligação estimada, coeficiente de inibição e modo de ligação que puderam ser avaliados e comparados dados de referência (HOWLETt 2002).
RESULTADOS E DISCUSSÃO:Obtemos modelos C1 e CB2 com o programa Modeller e verificado a qualidade do modelo com o programa PROCHECK, que mostra que apenas poucos resíduos (1,8 % CB1 e 0,6 % CB2) estão em regiões desfavoráveis para o gráfico Ramachandran. Com a simulação de docking obtemos para CB1 Ligante 1, energia de ligação E= -9.09 kcal/mol e coeficiente de inibição Ki = 217.43 nM (nanomolar), interagindo com os resíduos Ser88 (ligação de hidrogênio de 1,82 A),Gly99, Trp241, Phe191; o Ligante 2 com a proteína CB2, E= -9.79 kcal/mol e Ki = 66.40 nM (nanomolar) interagindo com os resíduos tyr347, Leu76, Glu327 (1,97 A) e Lys219 (1,99 A); nos dois caso podemos verificar a possibilidade de interação com os resíduos do sítio de ligação conforme previsto pelo programa LIGSITE (Leis, Schneider ; Zacharias, 2010), os quais podem ser visto nas figuras 1 e 2 em anexo. Forma prevista do modo de ligação do Ligante 1, no sítio de ligação da proteína CB1.
Figura 2
Forma prevista do modo de ligação do Ligante 2, no sítio de ligação da proteína CB2.
CONCLUSÕES:Segundo os dados da literatura (HOWLWETT 2002), (MONTERO, et al, 2010) os Ligante 1 e 2 tem as melhores potencialidades dentro do conjunto de moléculas testadas, com relação aos valores de coeficiente de inibição estimados e com as características relacionadas a seletividade em relação aos receptores,característica desejável (DA SILVA; SILVA, 2006) visto que os ligantes 1 e 2 deve ter afinidade por um receptor canabinóide e pouca pelo outro. Assim o Ligante 1 teve E= -8.16 kcal/mol e Ki = 1.05 uM (micromolar); já para o Ligante 2 com CB, tivemos E= -8.18 kcal/mol e Ki = 1.01 uM (micromolar).
AGRADECIMENTOS:
REFERÊNCIAS BIBLIOGRÁFICA:DA SILVA, .V. ; SILVA H.T.P Revista eletrônica de farmácia Vol. IV (1), 15-26, 2007
SANTOS-FILHO, O. A. DEALENCASTRO, R. B Quim. Nova, Vol. 26, No. 2, 253-259, 2003
SIMON LEIS, SEBASTIAN SCHENIDER AND MARTIN ZACHARIAS In Silico Prediction of Binding Sites on Proteins;. Current Medicinal Chemistry, 2010, 17, 1550-1562
MONTEIRO, C; CAMPILLO,N. E.; GOYA. European Journal of Medicinal Chemistry 40 (2005) 75–83
SALI, A.; BLUNDELL, T. L. Comparative protein modeling by satisfaction of spatial restraints. Journal of Molecular Biology. v. 234, p. 779-815, 1993.
HOWLETT et al (2002) Pharmacol.Rev. 54 161.
Fonte: http://www.abq.org.br/entequi/2012/trabalhos/50/50-2-9867.html

SOFTWARE MODELLER – MODELAGEM POR HOMOLOGIA
Sobre MODELLER
O software é usado para homologia ou modelagem comparativa de estruturas tridimensionais de proteínas (1,2). O usuário fornece um alinhamento de uma seqüência a ser modelada com estruturas relacionadas conhecidas e MODELLER calcula automaticamente um modelo contendo todos os átomos não-hidrogênio. MODELLER implementa modelagem comparativa de estrutura protéica por meio da satisfação de restrições espaciais (3,4) e pode realizar muitas tarefas adicionais, incluindo modelagem de novo de loops em estruturas de proteínas, otimização de vários modelos de estrutura proteica em relação a uma função objetiva definida flexivelmente Alinhamento múltiplo de sequências de proteínas e / ou estruturas, agrupamento, pesquisa de bases de dados de sequências, comparação de estruturas de proteínas, etc. MODELLER está disponível para download na maioria dos sistemas Unix / Linux, Windows e Mac.
Várias interfaces gráficas para MODELLER estão comercialmente disponíveis . Há também muitos outros recursos e pessoas usando o Modeller em interfaces gráficas ou da web ou outras estruturas.
- B. Webb, A. Sali. Modelagem Comparativa de Estrutura Protéica Usando o Modeller. Current Protocols in Bioinformatics 54, John Wiley & Sons, Inc., 5.6.1-5.6.37, 2016.
- MA Marti-Renom, A. Stuart, A. Fiser, R. Sánchez, F. Melo, A. Sali. Modelagem comparativa da estrutura protéica de genes e genomas. Annu Rev. Biophys. Biomol Struct. 29, 291-325, 2000.
- A. Sali e TL Blundell. modelagem de proteína comparativa pela satisfação de restrições espaciais. J. Mol. Biol. 234, 779-815, 1993.
- A. Fiser, RK Do e A. Sali. Modelagem de alças em estruturas de proteínas, Protein Science 9. 1753-1773, 2000.
A versão atual do Modeller é 9.21 , lançada em 11 de dezembro de 2018. Atualmente, o Modeller é mantido por Ben Webb .
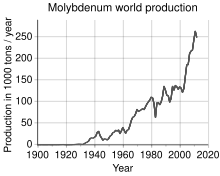
Molibdênio
O molibdênio (português brasileiro) ou molibdénio (português europeu) é um elemento químico de símbolo Mo e número atômico 42. O nome é derivado do neo-latim Molybdaenum, do grego antigo Μόλυβδος molybdos que significa chumbo, uma vez que seus minérios eram confundidos com os de chumbo.[1] Minerais de molibdênio são conhecidos ao longo da história, mas o elemento só foi descoberto (no sentido de diferenciação como uma nova entidade a partir dos sais minerais de outros metais) em 1778 por Carl Wilhelm Scheele. O metal foi isolado pela primeira vez em 1781 por Peter Jacob Hjelm.
O molibdênio não é encontrado naturalmente como um metal livre na Terra, mas em vários estados de oxidação em minerais. O elemento livre, que é um metal de cor prateada, tem o sexto maior ponto de fusão de todos os elementos conhecidos. Forma prontamente ligas estáveis e duras com o carbono, e por esta razão a maior parte da produção mundial do elemento (aproximadamente 80%) é na fabricação do aço, incluindo ligas resistentes e superligas.
A maioria dos compostos tem baixa solubilidade em água, porém o íon molibdato MoO2−
4 e é formado quando minerais contendo molibdênio entram em contato com o oxigênio e água. Industrialmente, compostos químicos de molibdênio (aproximadamente 14% da produção mundial), são usados em aplicações em alta temperatura e pressão, como pigmentos e catalisadores.
Enzimas contendo molibdênio são de longe a forma mais comum de catálise utilizada por algumas bactérias para quebrar a ligação química do nitrogênio molecular atmosférico, permitindo a fixação do nitrogênio. Pelo menos 50 enzimas com molibdênio são conhecidas em bactérias e animais, embora apenas algumas bactérias e cianobactérias estão envolvidas na fixação do nitrogênio. Estas nitrogenases contém molibdênio em uma forma diferente de outras enzimas, as quais contém molibdênio completamente oxidado incorporado em um cofator de molibdênio. Devido as diversas funções que várias enzimas com cofator de molibdênio, o elemento é requerido na dieta diária para todos os organismos eucariotas superiores, todavia não é requerido por todas as bactérias.
Propriedades físicas
O molibdênio é um metal de transição de coloração branco prateado e com uma dureza de 5,5 na escala de Mohs; além disso, tem um ponto de fusão de 2163ºC, o sexto maior entre os elementos naturais. Tem um dos menores coeficientes de expansão térmica entre os metais utilizados comercialmente,[2] e sua resistência à tração em fios aumenta três vezes, de aproximadamente 10 para 30 GPa, quando o diâmetro é reduzido para ~50–100 nm para 10 nm.[3]
Compostos e química
| Oxidação Estado |
Example[4] |
|---|---|
| −2 | Na2[Mo2(CO)10] |
| 0 | Mo(CO)6 |
| +1 | Na[C6H6Mo] |
| +2 | MoCl2 |
| +3 | Na3[Mo(CN)]6 |
| +4 | MoS2 |
| +5 | MoCl5 |
| +6 | MoF6 |

Estrutura de Keggin do ânion fosfomolibdato (P[Mo12O40]3−), um exemplo de polioxometalato
O molibdênio é um metal de transição com uma eletronegatividade de 2,16 na escala de Pauling e tem uma massa atômica padrão de 95,95 g/mol.[5][6] Não reage de modo visível com o oxigênio ou água a temperatura ambiente, e grande parte da oxidação ocorre em temperaturas superiores a 600 °C, resultando no trióxido de molibdênio:
- 2 Mo + 3 O2 → 2 MoO3
O trióxido é volátil e sublima em altas temperaturas. Isto previne a formação de uma camada protetora de óxido, que poderia impedir a oxidação do metal.[7] O molibdênio possui vários estados de oxidação, sendo o mais estável o +4 e +6 (em negrito na tabela). A química e os compostos demonstram mais similaridade com o tungstênio do que com o cromo. Um exemplo é a instabilidade dos compostos de molibdênio (III) e tungstênio (III) comparados a estabilidade dos compostos de cromo (III). O maior estado de oxidação é comum no óxido de molibdênio (VI), ao passo que o composto sulfurado normal é o dissulfeto de molibdênio MoS2.[8]
O óxido de molibdênio (VI) é solúvel em bases fortes alcalinas aquosas, formando molibdatos (MoO42−), que são oxidantes mais fracos que os cromatos mas demonstram uma tendência similar a formar oxiânions complexos pela condensação em valores de pH baixos, tais como [Mo7O24]6− and [Mo8O26]4−. Polimolibdatos podem incorporar outros íons em suas estruturas, formando polioxometalatos.[9] O heteropolimolibdato que contém fósforo, P[Mo12O40]3− é utilizado na espectroscopia do ultravioleta visível para a detecção do fósforo.[10] A faixa ampla de estados de oxidação do molibdênio é evidente na variedade de cloretos que forma:[8]
- Cloreto de Molibdênio (II) MoCl2 (amarelo)
- Cloreto de Molibdênio (III) MoCl3 (vermelho escuro)
- Tetracloreto de Molibdênio MoCl4 (preto)
- Cloreto de Molibdênio (V) MoCl5 (verde escuro)
- Cloreto de Molibdênio (VI) MoCl6 (marrom)
A estrutura do MoCl2 é composta de aglomerados de Mo6Cl84+ com quatro íons cloreto para compensar a carga.[8]
Assim como o cromo e outros metais de transição, o molibdênio pode formar ligações quádruplas, tais como no Mo2(CH3COO)4. Este composto pode ser transformado no Mo2Cl84−, que também tem uma ligação quádrupla.[8]
O estado de oxidação 0 é possível com o monóxido de carbono como ligante, tal como no molibdênio hexacarbonil, Mo(CO)6.[8]
História

Carl Wilhelm Scheele enunciou que a molibdenita continha um novo elemento químico.[11]
A Molibdenita – o principal minério do qual o molibdênio é extraído – era antigamente conhecida como Molibdena, que era confundida às vezes utilizada como grafite. A Molibdenita pode ser utilizada para escurecer a superfície ou como um sólido lubrificante, assim como o grafite.[12] Mesmo quando a Molibdenita era distinguível do grafite, era ainda confundida com o minério de chumbo galena; o nome do elemento veio do grego antigo Μόλυβδος molybdos, que significa chumbo.[2] Tem sido proposto que a palavra grega em si é uma palavra emprestada das línguas da região da Anatólia, atual Turquia.[13]
Apesar de uma liga de aço com molibdênio em uma espada japonesa do século XIV ter sido relatada, a técnica não foi amplamente empregada e posteriormente foi perdida.[14][15] Em 1754 no Ocidente, Bengt Andersson Qvist examinou a Molibdenita e constatou que ela não continha chumbo, e portanto não era o mesmo que a galena.[16]
Em 1778 o químico sueco Carl Wilhelm Scheele enunciou firmemente que a molibdenita não era de fato nem galena ou grafite.[11][17] Ao invéis disso, Scheele foi adiante e corretamente propôs que a Molibdenita era um minério de um novo elemento distinto, nomeado molybdenum para o mineral que o contém, e a partir do qual poderia ser isolado. Peter Jacob Hjelm isolou com sucesso o elemento a partir de uma redução com carbono e óleo de linhaça em 1781.[2][18]
Por aproximadamente um século após seu isolamento, o molibdênio não teve uso industrial em função de sua escassez relativa, dificuldade de extração do metal puro e a falta de maturidade das técnicas metalúrgicas apropriadas.[19][20][21] As primeiras ligas de aço com molibdênio demonstraram ser promissoras pelo aumento da dureza, mas esforços para manufaturá-las em larga escala foram travadas pelos resultados inconsistentes e a tendência de fragilidade e recristalização. Em 1906, William D. Coolidge solicitou uma patente para uma forma de molibdênio dúctil, para o seus uso em fornos de alta temperatura e como um suporte para o filamento de tungstênio em lâmpadas de bulbo; a oxidação e degradação requeriu que o molibdênio fosse fisicamente selado ou contido em um gás inerte.[22] Em 1913, Frank E. Elmore desenvolveu um processo de flotação para recuperar a molibdenita de minérios, método que permanece como o processo principal de isolamento do mineral.[23]
Durante a primeira Guerra Mundial, a demanda pelo elemento aumentou severamente; era usado tanto na blindagem de veículos quanto como um substituto para o tungstênio no aço rápido. Alguns tanques britânicos eram protegidos por 75 mm de blindagem de aço Hadfield porém isto se mostrou ineficiente. As blindagens foram substituídas com ligas de aço com molibdênio com 25 mm de espessura para aumento de velocidade dos veículos, maior facilidade de manobra e melhor proteção.[2] Os alemães também utilizaram molibdênio em ligas para a artilharia pesada. Isto foi porque o aço tradicional se fundia com o calor produzido para o lançamento de um projétil de uma tonelada.[24] Após a guerra, a demanda diminuiu até que avanços na metalurgia permitissem o desenvolvimento de aplicações de tempos de paz. Na Segunda Guerra, o molibdênio ganhou novamente importância estratégica como um substituto para o Tungstênio em ligas metálicas.[25]
Ocorrência e produção

Molibdenita no quartzo
O molibdênio é o 54º elemento mais abundante na crosta terrestre, e o 25º mais abundante nos oceanos, com uma concentração média de 10 partes por bilhão; e é o 42º elemento mais abundante no Universo.[2][26] A missão russa Luna 24 descobrindo um grão de molibdênio (1 × 0.6 µm) em um fragmento de piroxeno retirado da Mare Crisum na Lua.[27] A raridade relativa do molibdênio na crosta terrestre é compensada pela sua concentração em uma variedade de minérios insolúveis em água, Às vezes combinado com o enxofre, do mesmo modo que o cobre, que também é encontrado. Ainda que o elemento seja encontrado em minerais como a wulfenita (PbMoO4) e a Powellita(CaMoO4), a principal fonte comercial é a molibdenita (MoS2). O molibdênio é extraído com minério principal, e também é recuperado como um subproduto durante a extração do cobre e tungstênio.[1]
Historicamente a mina de Knaben, aberta em 1885 no sul da Noruega, foi a primeira dedicada a extração do molibdênio. Ficou fechada de 1973 a 2007, mas foi reaberta.[28] A produção mundial de molibdênio foi de 250 mil toneladas em 2011, sendo os maiores produtores a China (94.000 t), Estados Unidos (64.000 t), Chile (38.000 t), Peru (18.000 t) e México (12.000 t). As reservas mundiais estão estimadas em 10 milhões de toneladas, e em maior parte estão na China (4.3 Mt), Estados Unidos (2.7 Mt) e Chile (1.2 Mt). Por continente, 93% da produção é dividia em partes quase equivalentes entre América do Norte, América do Sul e China. A Europa e o resto da Ásia (principalmente Armênia, Rússia, Irã e Mongólia) produzem o remanescente.[29]
No processamento da molibdenita, ela primeiramente é aquecida até 700°C e o sulfeto é oxidado para o óxido de molibdênio (IV) pelo ar:[8]
- 2 MoS2 + 7 O2 → 2 MoO3 + 4 SO2
O minério oxidado é ou aquecido para 1100°C para sublimação, ou lixiviado com Amônia, que reage com o óxido para formar molibdatos solúveis em água:
- MoO3 + 2 NH4OH → (NH4)2(MoO4) + H2O
O cobre, uma impureza da molibdenita, é menos solúvel em amônia. Para a remoção completa da solução, é precipitado com sulfeto de hidrogênio.[8]
O molibdênio puro é produzido pela redução do óxido com hidrogênio, enquanto o molibdênio para a produção do aço é reduzido pela reação aluminotérmica com adição do ferro para produzir o ferromolibdênio, que comumente tem 60% de molibdênio.[8][30]
O molibdênio tem um valor aproximado de $30 mil por tonelada (preço de Agosto de 2009). Manteve um valor próximo de $10 mil por tonelada de 1977 até 2003, e alcançou, devido a um pico de demanda, um preço de $103 mil por tonelada em junho de 2005.[31] Em 2008, a London Metal Exchange anunciou que o elemento seria tratado como um commodity no câmbio.[32]
Aplicações
Ligas
Aproximadamente 86% da produção de molibdênio é utilizada em aplicações metalúrgicas tais como ligas, com o resto sendo usado como compostos em aplicações químicas, com 35% destinado a indústria de aços estruturais, 25% de aço inoxidável, 9% em aços rápidos e ferramentas, 6% em ferro fundido, 6% na forma do metal e 5% em superligas.[33]
A capacidade do molibdênio de resistir a temperaturas extremas sem expandir significativamente ou amolecer o torna útil em aplicações que envolvem calor intenso, incluindo fabricação de blindagens, partes de aeronaves, contatos elétricos, motores industriais e filamentos.[2][34][35]
A maioria das ligas de aço de alta resistência (por exemplo aços 41xx) contém de 0,25% a 8% de molibdênio.[1] Apesar das pequenas proporções, mais de 43 mil toneladas são usadas como um agente ligante a cada ano em aços inoxidáveis, ferramentas em aço, ferro fundido e superligas para alta temperatura.[26]
O molibdênio também é usado em ligas de aço devido a sua alta resistência à corrosão[36] e soldabilidade.[26][29]
Por causa de sua baixa densidade e preço mais estável, o molibdênio é algumas vezes utilizado no lugar do tungstênio.[26] Um exemplo é a série M de aços rápidos tais como M2, M4 e M42 em substituição a série T de aços rápidos, que contém tungstênio. O molibdênio pode ser implementando como um agente ligante e como uma cobertura de resistência a chama para outros metais. Embora o ponto de fusão seja 2623 ºC, o elemento rapidamente se oxida a temperatura superiores a 760 ºC tornando mais adequado para uso em ambiente com vácuo.[34]
TZM (Mo (~99%), Ti (~0.5%), Zr (~0.08%) e algum C) é uma superliga resistente a corrosão que resiste a sais de fluoretos fundidos a temperaturas superiores a 1300 ºC. Tem aproximadamente duas vezes a resistência do molibdênio puro, e é mais dútil e soldável, ademais em testes resistiu a corrosão de um sal eutético padrão e vapores de sais usados em reatores de sais fundidos por 1100 horas com pouca corrosão de difícil medição.[37][38]
Outras ligas baseadas em molibdênio e que não contém ferro têm aplicações limitadas. Por exemplo, por causa da resistência a corrosão contra o zinco fundido, ambos molibdênio puro e liga molibdênio/tungstênio (70%/30%) são usadas em tubulações, agitadores e bombas que estão em contato com o zinco fundido.[39]
Outras aplicações com o elemento puro
O pó do molibdênio é utilizado como um fertilizante para algumas plantas, tais como a couve-flor.[26] Na forma elemental também é utilizado em analisadores de NO, NO2, NOx para controle da poluição em plantas elétricas. A 350 ºC o elemento age como um catalisador para NO2/NOx para formar somente moléculas NO para leituras no infravermelho.[40]Anodos de molibdênio substituem o tungstênio em certas fontes de raio-X em baixa voltagem, para usos especializados como em mamografias.[41] O isótopo radioativo Molibdênio-99 é utilizado para gerar o Tecnécio-99m, que é utilizado para a imagiografia médica. O isótopo é armazenado e manuseado na forma de molibdatos.[42][43]
Compostos
O dissulfeto de molibdênio (MoS2) é usado como um lubrificante sólido e agente anti-desgastante em altas pressões e temperaturas. Ele forma um filme resistente na superfície metálica e é um aditivo comum para graxas (em alta temperatura e pressão) – no caso de uma falha catastrófica da graxa, uma camada fina de molibdênio previne o contato das partes lubrificadas.[44] Também possui propriedades semicondutoras como vantagens distintas sobre o tradicional silicone ou aplicações eletrônicas com o grafeno.[45] MoS2também é usado como um cataliSador no hidrocraqueamento de frações do petróleo que contém nitrogênio, enxofre e oxigênio.[46]
Outros compostos, incluem:
- Dissiliceto de molibdênio (MoSi2): cerâmica condutora elétrica com uso principal em elementos aquecedores operando em temperaturas superiores a 1500 °C em ar.[47]
- Trióxido de molibdênio (MoO3): usado como um adesivo entre metais e esmaltes. O Molibdato de Chumbo co-precipitado com cromato de chumbo e sulfato de chumbo é um pigmento laranja brilhante usado em cerâmicas e plásticos.[48] Os catalisadores de óxidos mistos à base de molibdénio são utilizados para reacções de oxidação selectivas. As aplicações típicas são a oxidação de propano, propileno ou acroleína em ácido acrílico.[49][50][51][52]
Papel biológico
Bioquímica
Nitrogenases. O papel mais importante do Molibdênio em seres vivos é como um heteroátomo em sítios atívos de certas enzimas.[53] Na fixação do nitrogênio em certas bactérias,[54][55] a enzima nitrogenase, que é envolvida na etapa final da redução do nitrogênio molecular, normalmente contém o molibdênio no sítio ativo embora a substituição deste por ferro e vanádio também seja conhecida.[56]
A reação que a enzima nitrogenase realiza é:
{\displaystyle \mathrm {N_{2}+8\ H^{+}+8\ e^{-}+16\ ATP+16\ H_{2}O\longrightarrow 2\ NH_{3}+H_{2}+16\ ADP+16\ P_{i}} }
Com prótons e elétrons da cadeia de transporte de eléltros, o nitrogênio é reduzido a amônia e gás hidrogênio livre. Este é um processo que requer energia, exigindo a hidrólise do ATP no ADP mais um fosfato livre (Pi).
Em 2008, foi evidenciado que a escassez de molibdênio nos oceanos primitivos foi um fator limitante por aproximadamente dois bilhões de anos até a posterior evolução da vida eucariota (que incluem todas as plantas e animais) pois estas não conseguem fixar o nitrogênio, e portanto devem adquirir a maior parte do nitrogênio oxidado adequado para a criação de compostos orgânicos nitrogenados, ou os próprios organismos (como proteínas) a partir de bactérias procariotas.[57][58][59] A escassez de molibdênio resultoru em uma falta relativa de oxigênio nos oceanos primitivos. A maior parte dos compostos de molibdênio tem baixa solubilidade em água, mais o íon molibdato MoO42− é solúvel e é formado quando minerais contendo molibdênio entram em contato com o oxigênio e água. Uma vez que o oxigênio feito pela vida primitiva foi produzido na água do mar, ajudou a dissolver o molibdênio em molibdatos solúveis a partir dos minerais no fundo do oceano, tornando possível pela primeira vez a fixação do nitrogênio por bactérias, e permitindo a elas prover mais compostos de nitrogênio fixado utilizáveis por formas superiores de vida. Em 2013, foi sugerida a possibilidade que o boro e o molibdênio catalisaram a produção do RNA em Marte com a vida sendo transferida para a terra por um meteorito por volta de três bilhões de anos atrás.[60]
Apesar do oxigênio tivesse promovido a fixação do nitrogênio ao tornar o molibdênio solúvel em água, também envenena diretamente as enzimas de nitrogenase. Assim na história antiga da Terra, após o oxigênio ter alcançado largas quantidades na água e no ar, organismos que continuaram a fixar o nitrogênio em condições aeróbicas precisaram isolar e proteger suas enzimas fixadoras de nitrogênios em heterocistos, ou estruturas protetoras similares do oxigênio disponível. Esta isolação estrutural nas reações de fixação do nitrogênio em organismos aeróbicos continua até a atualidade.

O cofator de molibdênio (imagem) é composto de uma molécula orgânica complexa chamada molibdopterina, que se liga a um átomo de molibdênio (VI) oxidado através do átomo de enxofre adjacente (ou ocasionalmente o selênio). Exceto pelas nitrogenases primitivas, todas as enzimas de molibdênio conhecidas usam este cofator.
Enzimas com cofator de molibdênio. Embora o molibdênio forme compostos com várias moléculas orgânicas, incluindo carboidratos e aminoácidos, ele é transportado através do corpo humano como um molibdato MoO42−.[61] Até 2002, pelo menos 50 enzimas contendo molibdênio eram conhecidas, a maior parte em bactérias, e seu número aumenta a cada ano;[62][63] tais enzimas incluem a aldeído oxidase, sulfito oxidase e a xantina oxidase.[2] Em alguns animais, e em humanos, a oxidação da xantina em ácido úrico, um processo do catabolismo da purina, é catalisada pela xantina oxidase. A atividade da xantina oxidase é diretamente proporcional a quantidade de molibdênio no organismo. Entretanto, uma concentração muito alta do elemento pode reverter a tendência e agir como um inibidor tanto no catabolismo da purina quanto em outros processos. A concentração de molibdênio também afeta síntese proteica, o metabolismo e o crescimento.[61]
Em animais e plantas um composto tricíclico chamado molibdopterina (que, apesar do nome, não contém o molibdênio) reage com o molibdato para formar um cofator contendo molibdênio denominado cofator de molibdênio. Protegidas da nitrogenases da filogenética primitiva, que incluem a fixação do nitrogênio em algumas bactérias e cianobactérias, todas as enzimas contendo molibdênio identificadas até a atualidade na natureza utilizam o cofator de molibdênio, no qual o elemento está no estado oxidativo VI, o mesmo que o do molibdato.[64] Enzimas de molibdênio em plantas e animais podem catalisar a oxidação e algumas vezes a redução de certas moléculas pequenas, como parte da regulação do ciclo do nitrogênio, ciclo do enxofre e ciclo do carbono.[65]
Ingestão diária e deficiência
O molibdênio é um elemento químico essencial para a vida dos animais.[66] Quatro enzimas com dependência de molibdênio em mamíferos são conhecidas, todas baseadas na pterina no cofator de molibdênio do sítio ativo: sulfito oxidase, xantina oxidase e aldeído oxidase.[67] Pessoas com deficiência em molibdênio tem um funcionamento deficiente da sulfito oxidase e são propensas a reações tóxicas de sulfitos nos alimentos.[68][69] O corpo humano contém aproximadamente 0,07 mg de molibdênio por quilograma do peso corporal.[70] Ocorrem em concentrações superiores no fígado e rins, e em concentrações menores nas vértebras.[26] O elemento também está presente dentro do esmalte dentárioe pode ajudar a prever sua queda.[71]
A ingestão diária de molibdênio varia de 0,12 a 0,24 mg, dependendo do teor de molibdênio no alimento.[72] Carnes de suínos, ovinos e bifes de fígado tem aproximadamente 1,5 partes por milhão de molibdênio. Outras fontes relevantes incluem feijões verdes, ovos, sementes de girassol, farinha de trigo, lentilhas, pepinos e cereais em grãos.[2] A toxicidade aguda não foi observada em humanos e depende fortemente do estado químico do elemento. Estudos com ratos demonstram uma dose letal mediana (LD50) menor que 180 mg/kg para alguns compostos.[73] Embora dados sobre a toxicidade em humanos não estejam disponíveis, estudos com animais têm demonstrado que a ingestão crônica de mais de 10 mg/dia pode causar diarreia, retardo no crescimento, infertilidade, baixo peso de recém-nascidos e gota; também pode afetar os pulmões, rins e o fígado.[72][74] O tungstato de sódio é um inibidor competitivo do molibdênio e dietas com tungstênio reduzem a concentração do molibdênio em tecidos.[26]
A deficiência de molibdênio na dieta a partir baixa concentração no solo tem sido associada com um aumento da taxa de câncer de esôfago em parte do norte da china até o Irão.[75][76] Comparados com os Estados Unidos, onde existe um grande suprimento do elemento no solo, pessoas nestas áreas deficientes têm um risco 16 vezes maior de desenvolver câncer esofágico e carcinoma de células escamosas.[77][carece de fontes]
A deficiência no elemento também tem sido relatada como uma consequência da nutrição parenteral total (alimentação intravenosa completa) por longos períodos de tempo. Resulta em altos níveis sanguíneos de sulfitos e ácido úrico, quase do mesmo modo que a deficiência no cofator de molibdênio. Todavia, uma vez que presumivelmente a deficiência em molibdênio a partir deste mecanismo é observada primariamente em adultos, as consequências neurológicas não têm sido sinalizadas como para a deficiência de um fator congênito.[78]
Doenças relacionadas
Uma doença congênita da deficiência do cofator de molibdênio, observada em crianças, resulta na interferência da capacidade do organismo de usar o molibdênio em enzimas. Causa altos níveis de sulfitos e ácido úrico, e danos neurológicos.[79][80]
Antagonismo Cobre-Molibdênio
Altos níveis de molibdênio podem interferir na captação do Cobre, provocando a deficiência de cobre. O molibdênio previne proteínas de se ligar ao cobre, e também aumentam a quantidade de cobre que é excretado na urina. Ruminantes que consumem grandes quantidades de molibdênio desenvolvem sintomas que incluem diarreia, crescimento atrofiado, anemia e perda do pigmento do cabelo. Estes sintomas podem ser aliviados pela administração de mais cobre no organismo, tanto na forma de injeção quanto por uma dieta regulada.[81] A condição, como uma efetiva deficiência de cobre, pode ser agravada por excesso de Enxofre.[26][82]
A redução ou deficiência de cobre também pode ser deliberadamente reduzida para propósitos terapêuticos pelo composto tetratiomolibdato de amónio, no qual o ânion vermelho brilhante de tetratiomolibdato é um agente quelante do cobre. O composto foi usado pela primeira vez com este propósito para no tratamento da toxicose de cobre em animais. Foi então introduzido como forma de tratamento para doença de Wilson, uma desordem metabólica do cobre que é hereditária em humanos; o composto age tanto pela competição na absorção do cobre no intestino e no aumento da excreção do elemento. Também foi descoberto que tem efeito inibidor na angiogênese, potencialmente pela inibição do íon cobre do processo de translocação da membrana envolvendo um caminho de secreção não-clássico.[83] Isto é um tratamento investigatório interessante para o câncer, degeneração macular relacionada a idade e outras doenças com característica deposição no sangue.[84][85]
Isótopos
O molibdênio tem 6 isótopos estáveis e cerca de duas dezenas de radioisótopos, a maior parte com tempos de vida média da ordem de segundos. O 99Mo se usa em geradores de 99Mo / 99mTc para a indústria de isótopos nucleares. Estima-se que este mercado de produtos de 99Tc mova aproximadamente 100 milhões de euros ao ano.
Referências
- ↑ Ir para:a b c Lide, David R., ed. (1994). «Molybdenum». CRC Handbook of Chemistry and Physics. 4. [S.l.]: Chemical Rubber Publishing Company. p. 18. ISBN 0-8493-0474-1
- ↑ Ir para:a b c d e f g h Emsley, John (2001). Nature’s Building Blocks. Oxford: Oxford University Press. pp. 262–266. ISBN 0-19-850341-5
- ↑ Shpak, Anatoly P.; Kotrechko, Sergiy O.; Mazilova, Tatjana I; Mikhailovskij, Igor M (2009). «Inherent tensile strength of molybdenum nanocrystals». Science and Technology of Advanced Materials. 10 (4): 045004. Bibcode:2009STAdM..10d5004S. doi:10.1088/1468-6996/10/4/045004
- ↑ Schmidt, Max (1968). «VI. Nebengruppe». Anorganische Chemie II. (em German). [S.l.]: Wissenschaftsverlag. pp. 119–127
- ↑ M. E. Wieser; M. Berglund (2009). «Atomic weights of the elements 2007 (IUPAC Technical Report)» (PDF). Pure and Applied Chemistry. 81 (11): 2131–2156. doi:10.1351/PAC-REP-09-08-03
- ↑ Meija, J.; et al. (2013). «Current Table of Standard Atomic Weights in Alphabetical Order: Standard Atomic weights of the elements». Consultado em 13 de março de 2015. Arquivado do original em 29 de abril de 2014
- ↑ Davis, Joseph R. (1997). Heat-resistant materials. Molybdenum. [S.l.]: ASM International. p. 365. ISBN 0-87170-596-6
- ↑ Ir para:a b c d e f g h Holleman, Arnold F.; Wiberg, Egon; Wiberg, Nils (1985). Lehrbuch der Anorganischen Chemie 91–100 ed. [S.l.]: Walter de Gruyter. pp. 1096–1104. ISBN 3-11-007511-3
- ↑ Pope, Michael T.; Müller, Achim (1997). «Polyoxometalate Chemistry: An Old Field with New Dimensions in Several Disciplines». Angewandte Chemie International Edition. 30: 34. doi:10.1002/anie.199100341
- ↑ Nollet, Leo M. L., ed. (2000). Handbook of water analysis. New York, NY: Marcel Dekker. pp. 280–288. ISBN 978-0-8247-8433-1
- ↑ Ir para:a b Gagnon, Steve. «Molybdenum». Jefferson Science Associates, LLC. Consultado em 6 de maio de 2007
- ↑ Lansdown, A. R. (1999). Molybdenum disulphide lubrication. Tribology and Interface Engineering. 35. [S.l.]: Elsevier. ISBN 978-0-444-50032-8
- ↑ Melchert, Craig. «Greek mólybdos as a Loanword from Lydian» (PDF). University of North Carolina at Chapel Hill. Consultado em 23 de abril de 2011
- ↑ International Molybdenum Association, “Molybdenum History” Arquivado em 22 de julho de 2013, no Wayback Machine.
- ↑ Institute, American Iron and Steel (1948). Accidental use of molybdenum in old sword led to new alloy. [S.l.: s.n.]
- ↑ Van der Krogt, Peter (10 de janeiro de 2006). «Molybdenum». Elementymology & Elements Multidict. Consultado em 20 de maio de 2007
- ↑ Scheele, C. W. K. (1779). «Versuche mit Wasserbley;Molybdaena». Svenska vetensk. Academ. Handlingar. 40: 238
- ↑ Hjelm, P. J. (1788). «Versuche mit Molybdäna, und Reduction der selben Erde». Svenska vetensk. Academ. Handlingar. 49: 268
- ↑ Hoyt, Samuel Leslie (1921). Metallography, Volume 2. [S.l.]: McGraw-Hill
- ↑ Krupp, Alfred; Wildberger, Andreas (1888). The metallic alloys: A practical guide for the manufacture of all kinds of alloys, amalgams, and solders, used by metal-workers … with an appendix on the coloring of alloys. [S.l.]: H.C. Baird & Co. p. 60
- ↑ Gupta, C.K. (1992). Extractive Metallurgy of Molybdenum. [S.l.]: CRC Press. ISBN 978-0-8493-4758-0
- ↑ Reich, Leonard S (22 de agosto de 2002). The Making of American Industrial Research: Science and Business at Ge and Bell, 1876–1926. [S.l.: s.n.] p. 117. ISBN 9780521522373
- ↑ Vokes, Frank Marcus (1963). Molybdenum deposits of Canada. [S.l.: s.n.] p. 3
- ↑ Sam Kean. The Disappearing Spoon. Page 88–89
- ↑ Millholland, Ray (agosto de 1941). «Battle of the Billions: American industry mobilizes machines, materials, and men for a job as big as digging 40 Panama Canals in one year». Popular Science: 61
- ↑ Ir para:a b c d e f g h Considine, Glenn D., ed. (2005). «Molybdenum». Van Nostrand’s Encyclopedia of Chemistry. New York: Wiley-Interscience. pp. 1038–1040. ISBN 978-0-471-61525-5
- ↑ Jambor, J.L.; et al. (2002). «New mineral names» (PDF). American Mineralogist. 87: 181
- ↑ Langedal, M (1997). «Dispersion of tailings in the Knabena—Kvina drainage basin, Norway, 1: Evaluation of overbank sediments as sampling medium for regional geochemical mapping». Journal of Geochemical Exploration. 58 (2–3): 157. doi:10.1016/S0375-6742(96)00069-6
- ↑ Ir para:a b «Molybdenum Statistics and Information». U.S. Geological Survey. 10 de maio de 2007. Consultado em 10 de maio de 2007
- ↑ Gupta, C. K. (1992). Extractive Metallurgy of Molybdenum. [S.l.]: CRC Press. pp. 1–2. ISBN 978-0-8493-4758-0
- ↑ «Dynamic Prices and Charts for Molybdenum». InfoMine Inc. 2007. Consultado em 7 de maio de 2007
- ↑ «LME to launch minor metals contracts in H2 2009». London Metal Exchange. 4 de setembro de 2008. Consultado em 28 de julho de 2009. Arquivado do original em 6 de maio de 2009
- ↑ Pie chart of world Mo uses Arquivado em 10 de março de 2012, no Wayback Machine.. London Metal Exchange.
- ↑ Ir para:a b «Molybdenum». AZoM.com Pty. Limited. 2007. Consultado em 6 de maio de 2007
- ↑ «Espacenet – Bibliographic data». worldwide.espacenet.com (em inglês). Consultado em 26 de março de 2017 horizontal tab character character in
|titulo=at position 13 (ajuda) - ↑ Neto, Pereira; Olímpio, Joaquim; Silva, Raphael Oliveira da; Silva, Edilaine Honório da; Moreto, Jeferson Aparecido; Bandeira, Rafael Marinho; Manfrinato, Marcos Dorigão; Rossino, Luciana Sgarbi; Neto, Pereira (1 de dezembro de 2016). «Wear and Corrosion Study of Plasma Nitriding F53 Super duplex Stainless Steel». Materials Research. 19 (6): 1241–1252. ISSN 1516-1439. doi:10.1590/1980-5373-mr-2015-0656
- ↑ Smallwood, Robert E. (1984). «TZM Moly Alloy». ASTM special technical publication 849: Refractory metals and their industrial applications: a symposium. [S.l.]: ASTM International. p. 9. ISBN 9780803102033
- ↑ «Compatibility of Molybdenum-Base Alloy TZM, with LiF-BeF2-ThF4-UF4». Oak Ridge National Laboratory Report. Dezembro de 1969. Consultado em 2 de setembro de 2010
- ↑ Cubberly, W. H.; Bakerjian, Ramon (1989). Tool and manufacturing engineers handbook. [S.l.]: Society of Manufacturing Engineers. p. 421. ISBN 978-0-87263-351-3
- ↑ Lal, S.; Patil, R. S. (2001). «Monitoring of atmospheric behaviour of NOx from vehicular traffic». Environmental Monitoring and Assessment. 68 (1): 37–50. PMID 11336410. doi:10.1023/A:1010730821844
- ↑ Lancaster, Jack L. «Ch. 4: Physical determinants of contrast». Physics of Medical X-Ray Imaging (PDF). [S.l.]: University of Texas Health Science Center. Consultado em 27 de fevereiro de 2015. Arquivado do original (PDF) em 10 de outubro de 2015
- ↑ Theodore Gray. The Elements. pp. 105–107
- ↑ Gottschalk, A (1969). «Technetium-99m in clinical nuclear medicine». Annual review of medicine. 20 (1): 131–40. PMID 4894500. doi:10.1146/annurev.me.20.020169.001023
- ↑ Winer, W. (1967). «Molybdenum disulfide as a lubricant: A review of the fundamental knowledge». Wear. 10 (6): 422. doi:10.1016/0043-1648(67)90187-1
- ↑ «New transistors: An alternative to silicon and better than graphene». Physorg.com. 30 de janeiro de 2011. Consultado em 30 de janeiro de 2011
- ↑ Topsøe, H.; Clausen, B. S.; Massoth, F. E. (1996). Hydrotreating Catalysis, Science and Technology. Berlin: Springer-Verlag
- ↑ Moulson, A. J.; Herbert, J. M. (2003). Electroceramics: materials, properties, applications. [S.l.]: John Wiley and Sons. p. 141. ISBN 0-471-49748-7
- ↑ International Molybdenum Association. imoa.info.
- ↑ Kinetic studies of propane oxidation on Mo and V based mixed oxide catalysts (PDF). Tese de doutorado. [S.l.: s.n.] 2011
- ↑ «Surface chemistry of phase-pure M1 MoVTeNb oxide during operation in selective oxidation of propane to acrylic acid» (PDF). Journal of Catalysis. 285: 48-60. 2012. doi:10.1016/j.jcat.2011.09.012. Arquivado do original (PDF) em 30 de outubro de 2016
- ↑ «Multifunctionality of Crystalline MoV(TeNb) M1 Oxide Catalysts in Selective Oxidation of Propane and Benzyl Alcohol». ACS Catalysis. doi:10.1021/cs400010q
- ↑ «The reaction network in propane oxidation over phase-pure MoVTeNb M1 oxide catalysts» (PDF). Journal of Catalysis. 311: 369-385. 2014. doi:10.1016/j.jcat.2013.12.008. Arquivado do original (PDF) em 15 de fevereiro de 2016
- ↑ Mendel, Ralf R. (2013). «Chapter 15 Metabolism of Molybdenum». In: Banci, Lucia (Ed.). Metallomics and the Cell. Col: Metal Ions in Life Sciences. 12. [S.l.]: Springer. ISBN 978-94-007-5560-4. ISSN 1559-0836. doi:10.1007/978-94-007-5561-10_15 electronic-book ISBN 978-94-007-5561-1 electronic-issn 1868-0402
- ↑ Silva, Alinne da; Franzini, Vinícius I.; Piccolla, Cristiano D.; Muraoka, Takashi; Silva, Alinne da; Franzini, Vinícius I.; Piccolla, Cristiano D.; Muraoka, Takashi (1 de fevereiro de 2017). «Molybdenum supply and biological fixation of nitrogen by two Brazilian common bean cultivars». Revista Brasileira de Engenharia Agrícola e Ambiental. 21 (2): 100–105. ISSN 1415-4366. doi:10.1590/1807-1929/agriambi.v21n2p100-105
- ↑ Vieira, Rogerio Faria; Lima, Renan Cardoso; Prado, Adalgisa Leles do; Júnior, Trazilbo José de Paula; Soares, Bruno de Almeida (2 de setembro de 2016). «Split application of molybdic fertilizer at the reproductive stage of common bean increases the molybdenum content in seed». Acta Scientiarum. Agronomy (em inglês). 38 (4): 529–533. ISSN 1807-8621. doi:10.4025/actasciagron.v38i4.30536
- ↑ Dos Santos, Patricia C.; Dean, Dennis R. (2008). «A newly discovered role for iron-sulfur clusters». PNAS. 105 (33): 11589–11590. Bibcode:2008PNAS..10511589D. PMC 2575256
 . PMID 18697949. doi:10.1073/pnas.0805713105
. PMID 18697949. doi:10.1073/pnas.0805713105 - ↑ Scott, C.; Lyons, T. W.; Bekker, A.; Shen, Y.; Poulton, S. W.; Chu, X.; Anbar, A. D. (2008). «Tracing the stepwise oxygenation of the Proterozoic ocean». Nature. 452 (7186): 456–460. Bibcode:2008Natur.452..456S. PMID 18368114. doi:10.1038/nature06811
- ↑ «International team of scientists discover clue to delay of life on Earth». Eurekalert.org. Consultado em 25 de outubro de 2008
- ↑ «Scientists uncover the source of an almost 2 billion year delay in animal evolution». Eurekalert.org. Consultado em 25 de outubro de 2008
- ↑ «Primordial broth of life was a dry Martian cup-a-soup». New Scientist. 29 de agosto de 2013. Consultado em 29 de agosto de 2013
- ↑ Ir para:a b Mitchell, Phillip C. H. (2003). «Overview of Environment Database». International Molybdenum Association. Consultado em 5 de maio de 2007. Arquivado do original em 18 de outubro de 2007
- ↑ Enemark, John H.; Cooney, J. Jon A.; Wang, Jun-Jieh; Holm, R. H. (2004). «Synthetic Analogues and Reaction Systems Relevant to the Molybdenum and Tungsten Oxotransferases». Chem. Rev. 104 (2): 1175–1200. PMID 14871153. doi:10.1021/cr020609d
- ↑ Mendel, Ralf R.; Bittner, Florian (2006). «Cell biology of molybdenum». Biochimica et Biophysica Acta. 1763 (7): 621–635. PMID 16784786. doi:10.1016/j.bbamcr.2006.03.013
- ↑ Fischer, B; Enemark, JH; Basu, P (1998). «A chemical approach to systematically designate the pyranopterin centers of molybdenum and tungsten enzymes and synthetic models». Journal of Inorganic Biochemistry. 72 (1–2): 13–21. PMID 9861725. doi:10.1016/S0162-0134(98)10054-5. Summarized in MetaCyc Compound: molybdopterin. Accessed Nov. 16, 2009.
- ↑ Kisker, C.; Schindelin, H.; Baas, D.; Rétey, J.; Meckenstock, R.U; Kroneck, P.M.H (1999). «A structural comparison of molybdenum cofactor-containing enzymes». FEMS Microbiol. Rev. 22 (5): 503–521. PMID 9990727. doi:10.1111/j.1574-6976.1998.tb00384.x
- ↑ Schwarz, Guenter; Belaidi, Abdel A. (2013). «Chapter 13. Molybdenum in Human Health and Disease». In: Astrid Sigel; Helmut Sigel; Roland K. O. Sigel. Interrelations between Essential Metal Ions and Human Diseases. Col: Metal Ions in Life Sciences. 13. [S.l.]: Springer. pp. 415–450. doi:10.1007/978-94-007-7500-8_13
- ↑ Mendel, Ralf R. (2009). «Cell biology of molybdenum». BioFactors. 35 (5): 429–34. PMID 19623604. doi:10.1002/biof.55
- ↑ Blaylock Wellness Report, February 2010, page 3.
- ↑ Cohen, HJ; Drew, RT; Johnson, JL; Rajagopalan, KV (1973). «Molecular Basis of the Biological Function of Molybdenum. The Relationship between Sulfite Oxidase and the Acute Toxicity of Bisulfite and SO2». Proceedings of the National Academy of Sciences of the United States of America. 70 (12 Pt 1–2): 3655–3659. Bibcode:1973PNAS…70.3655C. PMC 427300
 . PMID 4519654. doi:10.1073/pnas.70.12.3655
. PMID 4519654. doi:10.1073/pnas.70.12.3655 - ↑ Holleman, Arnold F.; Wiberg, Egon (2001). Inorganic chemistry. [S.l.]: Academic Press. p. 1384. ISBN 0-12-352651-5
- ↑ Curzon, M. E. J.; Kubota, J.; Bibby, B. G. (1971). «Environmental Effects of Molybdenum on Caries» (PDF). Journal of Dental Research. 50 (1): 74–77. doi:10.1177/00220345710500013401
- ↑ Ir para:a b Coughlan, M. P. (1983). «The role of molybdenum in human biology». Journal of Inherited Metabolic Disease. 6 (S1): 70–77. PMID 6312191. doi:10.1007/BF01811327
- ↑ «Risk Assessment Information System: Toxicity Summary for Molybdenum». Oak Ridge National Laboratory. Consultado em 23 de abril de 2008. Arquivado do original em 19 de setembro de 2007
- ↑ Barceloux, Donald G.; Barceloux, Donald (1999). «Molybdenum». Clinical Toxicology. 37(2): 231–237. PMID 10382558. doi:10.1081/CLT-100102422
- ↑ Yang, Chung S. (1980). «Research on Esophageal Cancer in China: a Review» (PDF). Cancer Research. 40 (8 Pt 1): 2633–44. PMID 6992989
- ↑ Nouri, Mohsen; Chalian, Hamid; Bahman, Atiyeh; Mollahajian, Hamid; et al. (2008). «Nail Molybdenum and Zinc Contents in Populations with Low and Moderate Incidence of Esophageal Cancer» (PDF). Archives of Iranian Medicine. 11: 392
- ↑ Taylor, Philip R.; Li, Bing; Dawsey, Sanford M.; Li, Jun-Yao; Yang, Chung S.; Guo, Wande; Blot, William J. (1994). «Prevention of Esophageal Cancer: The Nutrition Intervention Trials in Linxian, China» (PDF). Cancer Research. 54 (7 Suppl): 2029s–2031s. PMID 8137333. Consultado em 6 de março de 2015. Arquivado do original (PDF) em 4 de junho de 2016
- ↑ Abumrad, NN (1984). «Molybdenum—is it an essential trace metal?». Bulletin of the New York Academy of Medicine. 60 (2): 163–71. PMC 1911702
 . PMID 6426561
. PMID 6426561 - ↑ Smolinsky, B.; Eichler, S. A.; Buchmeier, S.; Meier, J. C.; Schwarz, G.; et al. (2008). «Splice-specific Functions of Gephyrin in Molybdenum Cofactor Biosynthesis». Journal of Biological Chemistry. 283 (25): 17370–9. PMID 18411266. doi:10.1074/jbc.M800985200
- ↑ Reiss, J. (2000). «Genetics of molybdenum cofactor deficiency». Human Genetics. 106(2): 157–63. PMID 10746556. doi:10.1007/s004390051023
- ↑ Suttle, N. F. (1974). «Recent studies of the copper-molybdenum antagonism». CABI Publishing. Proceedings of the Nutrition Society. 33 (3): 299–305. PMID 4617883. doi:10.1079/PNS19740053
- ↑ Hauer, Gerald Copper deficiency in cattle. Bison Producers of Alberta. Accessed Dec. 16, 2010.
- ↑ Nickel, W (2003). «The Mystery of nonclassical protein secretion, a current view on cargo proteins and potential export routes». Eur. J. Biochem. 270 (10): 2109–2119. PMID 12752430. doi:10.1046/j.1432-1033.2003.03577.x
- ↑ Brewer GJ; Hedera, P; Kluin, KJ; Carlson, M; Askari, F; Dick, RB; Sitterly, J; Fink, JK; et al. (2003). «Treatment of Wilson disease with ammonium tetrathiomolybdate: III. Initial therapy in a total of 55 neurologically affected patients and follow-up with zinc therapy». Arch Neurol. 60 (3): 379–85. PMID 12633149. doi:10.1001/archneur.60.3.379
- ↑ Brewer, GJ; Dick, RD; Grover, DK; Leclaire, V; Tseng, M; Wicha, M; Pienta, K; Redman, BG; Jahan, T; Sondak, VK; Strawderman, M; LeCarpentier, G; Merajver, SD (2000). «Treatment of metastatic cancer with tetrathiomolybdate, an anticopper, antiangiogenic agent: Phase I study». Clinical cancer research : an official journal of the American Association for Cancer Research. 6 (1): 1–10. PMID 10656425
Cientistas criam implante que produz palavras a partir de ondas cerebrais
O sistema do trato vocal virtual conecta-se diretamente ao cérebro e sintetiza fala a partir de sinais cerebrais associados com os aspectos mecânicos da fala, como movimentos da mandíbula, laringe, lábios e língua. Eventualmente, o dispositivo poderia ser usado por pessoas que perderam a capacidade de falar. A pesquisa foi publicada nesta quinta-feira (25) na renomada científica revista Nature.
CELLO – Automação de design de circuitos genéticos.
Circuito de programação para biologia sintética
À medida que as técnicas de biologia sintética se tornam mais poderosas, os pesquisadores estão antecipando um futuro em que o projeto de circuitos biológicos será semelhante ao projeto de circuitos integrados em eletrônica. Nielsen et al. descreve o que é essencialmente uma linguagem de programação para projetar circuitos computacionais em células vivas. Os circuitos gerados nos plasmídeos expressos em Escherichia coli requeriam isolamento cuidadoso de seu contexto genético, mas funcionavam primariamente conforme especificado. Os circuitos poderiam, por exemplo, regular as funções celulares em resposta a múltiplos sinais ambientais. Tal estratégia pode facilitar o desenvolvimento de circuitos mais complexos por engenharia genética.
Resumo estruturado
INTRODUÇÃO
As células respondem ao ambiente, tomam decisões, constroem estruturas e coordenam tarefas. Subjacentes a esses processos estão operações computacionais realizadas por redes de proteínas reguladoras que integram sinais e controlam o tempo de expressão gênica. Aproveitar essa capacidade é fundamental para projetos de biotecnologia que exigem tomada de decisões, controle, detecção ou organização espacial. Foi demonstrado que as células podem ser programadas usando circuitos genéticos sintéticos compostos de reguladores organizados para gerar uma operação desejada. No entanto, a construção de circuitos simples é demorada e pouco confiável.
JUSTIFICATIVA
A automação de projeto eletrônico (EDA) foi desenvolvida para auxiliar os engenheiros no projeto de eletrônicos baseados em semicondutores. Em um esforço para acelerar o projeto de circuitos genéticos, aplicamos princípios da EDA para permitir o aumento da complexidade do circuito e simplificar a incorporação da regulação gênica sintética em projetos de engenharia genética. Usamos a linguagem de descrição de hardware Verilog para permitir que um usuário descreva uma função de circuito. O usuário também especifica os sensores, os atuadores e o “arquivo de restrições do usuário” (UCF), que define o organismo, a tecnologia do gate e as condições operacionais válidas. Violoncelo ( www.cellocad.org) usa essa informação para projetar automaticamente uma seqüência de DNA que codifica o circuito desejado. Isso é feito por meio de um conjunto de algoritmos que analisam o texto Verilog, criam o diagrama de circuito, atribuem portas, equilibram restrições para construir o DNA e simulam o desempenho.
RESULTADOS
O violoncelo desenha circuitos desenhando uma biblioteca de portas lógicas booleanas. Aqui, a tecnologia de gate consiste de lógica NOT / NOR baseada em repressores. A conexão do gate é simplificada definindo os sinais de entrada e saída como fluxos de RNA polimerase (RNAP). Descobrimos que os portões precisam ser isolados de seu contexto genético para funcionar de forma confiável no contexto de diferentes circuitos. Cada porta é isolada usando terminadores fortes para bloquear o vazamento de RNAP, e a permutabilidade de entrada é melhorada usando ribozimas e espaçadores de promotores. Essas peças são variadas para cada porta para evitar quebras devido à recombinação. Medir a carga de cada porta e incorporar isso nos algoritmos de otimização reduz ainda mais a pressão evolutiva.
O violoncelo foi aplicado ao projeto de 60 circuitos para Escherichia coli , onde a função do circuito foi especificada usando o código Verilog e transformada em uma seqüência de DNA. As sequências de DNA foram construídas conforme especificado sem ajuste adicional, exigindo 880.000 pares de bases de montagem de DNA. Destes, 45 circuitos foram executados corretamente em cada estado de saída (até 10 reguladores e 55 partes). Em todos os circuitos, 92% dos 412 estados de saída funcionaram como previsto.
CONCLUSÃO
Nosso trabalho constitui uma linguagem de descrição de hardware para programação de células vivas. Isso exigiu o codesenvolvimento de algoritmos de design com portas que são suficientemente simples e robustas para serem conectadas por algoritmos automatizados. Demonstramos que os princípios de engenharia podem ser aplicados para identificar e suprimir erros que complicam as composições de sistemas maiores. Essa abordagem leva a uma genética altamente repetitiva e modular, em contraste com a codificação de redes regulatórias naturais. O uso de uma linguagem independente de hardware e a criação de UCFs adicionais permitirão que um único design seja transformado em DNA para diferentes organismos, terminais genéticos, condições de operação e tecnologias de gate.
Programação genética usando violoncelo.
Um usuário especifica a função de circuito desejada no código Verilog, e isso é transformado em uma sequência de DNA. Um circuito de exemplo é mostrado (0xF6); as curvas vermelha e azul são estados de saída preditos para populações de células, e as distribuições de preto sólido são dados de citometria de fluxo experimental. As saídas são mostradas para todas as combinações de estados do sensor; sinais de mais e menos indicam a presença ou ausência de sinal de entrada. RBS, local de ligao ao ribossoma; RPU, unidade promotora relativa; YFP, proteína fluorescente amarela
. Fig. 1 Visão geral do violoncelo.
Fig. 1 Visão geral do violoncelo.
( A ) Usuários de violoncelo escrevem código Verilog e selecionam ou carregam sensores e um UCF. Com base no design do Verilog, uma tabela de verdade é construída a partir da qual um diagrama de circuito é sintetizado. Reguladores são atribuídos a partir de uma biblioteca para cada porta (cada cor é um repressor diferente). O desenho combinatório é então usado para concatenar partes em uma sequência de DNA linear. O SBOL Visual ( 101 ) é usado para os símbolos das peças. Flechas levantadas são promotores, círculos em hastes são isolantes de ribozimas, hemisférios são ERBs, setas grandes são seqüências codificadoras de proteínas e “T” s são terminadores. As cores das peças correspondem aos portões físicos. ( B) A especificação física para a UCF Eco1C1G1T1. O circuito e os sensores são inseridos em um plasmídeo; o outro plasmídeo contém o promotor de saída do circuito, que pode ser usado para dirigir a expressão de uma proteína fluorescente ou outro atuador. Ambos os plasmídeos devem estar presentes na cepa especificada para que o projeto seja válido.
 Fig. 2 Atribuição de portões genéticos ao diagrama de circuito.
Fig. 2 Atribuição de portões genéticos ao diagrama de circuito.
( A ) Um conjunto de quatro portas baseadas em diferentes repressores (cores) conectados em várias permutações para construir diferentes funções de circuito. As entradas (A, B e C) são promotores de entrada do sensor; o promotor de saída do circuito (X) controla o gene de atuação. ( B ) As formas das funções de resposta da porta determinam se elas podem ser conectadas funcionalmente. O portão laranja (PhlF) tem um grande alcance dinâmico (linhas tracejadas) que atravessa o limiar do portão roxo (BetI). No entanto, na ordem inversa, os portões não se conectam funcionalmente. ( C) As relações combinatórias dos repressores da biblioteca de portas isoladas são mostradas nas posições a montante (porta A) e a jusante (porta B). A escala de cores à direita indica a mudança relativa (normalizada), calculada como a faixa máxima de saída que pode ser alcançada conectando a porta A ao gate B. Os números indicam diferentes RBSs. Os gráficos da esquerda e da direita mostram quando a porta A regula a posição 1 e a posição 2, respectivamente. Portões que são excluídos da posição 2 por causa do roadblock são mostrados em preto (fig. S9). ( D ) A probabilidade de encontrar um circuito funcional versus o número de portas lógicas. A probabilidade de um circuito funcional é definida como a probabilidade de uma atribuição aleatória passar na análise do limiar de entrada (fig. S31) e não possuir combinações de roadblocking. ( E) A convergência do algoritmo de atribuição de porta de recozimento simulado (fig. S28). Inserções de gráfico de barras: as barras pretas devem estar LIGADAS; as barras cinzas devem estar DESLIGADAS; o eixo y é a saída no RPU em uma escala de log e o eixo x é o estado de entrada (da esquerda para a direita: 000, 001, 010, 011, 110, 101, 110, 111). A pontuação do circuito ( S ) é definida como a razão entre o estado ON previsto mais baixo e o estado OFF previsto mais alto (fig. S26 e eq. S2). Um exemplo de pesquisa é mostrado para o diagrama de circuito nas inserções; as cores correspondem aos repressores atribuídos a cada porta ( Fig. 3B ).
Entradas correspondem à ausência ou presença de IPTG 1 mM (direita – / +) e aTc (2 ng / ml; esquerda – / +) ( 84 ). (B ) As arquiteturas dos portões isolados. Algumas portas têm várias versões com diferentes seqüências RBS. As seqüências de DNA do gate são fornecidas na tabela S8. ( C ) Um exemplo de uma função de resposta para uma porta NOT baseada no repressor PhlF. A mudança no limite para os três RBSs é mostrada. Os dados para todas as portas isoladas são mostrados na fig. S4 ( D ) O impacto de cada porta no crescimento celular como uma função da sua atividade de promotor de entrada. O crescimento celular foi medido como OD 600 e normalizada pelo crescimento do controlo não indutor de 6 horas após a indução ( 84 ). Os quatro portais que reduziram o crescimento em> 20% estão indicados. Barras de erro são um SD de crescimento celular normalizado [ eixo y em (D)] e a mediana [eixo y em (A) e (C); eixo x em (C) e (D)] para três experiências independentes realizadas no mesmo dia.
Continua em: http://science.sciencemag.org/content/352/6281/aac7341
NOVO PROCESSO DE PRODUÇÃO DE ÒXIDO DE GRAFENO À TEMPERATURA AMBIENTE
NOVO PROCESSO DE PRODUÇÃO DE ÒXIDO DE GRAFENO À TEMPERATURA
AMBIENTE
FRANCISLEI SANTA ANNA SANTOS1*
1 Msc. em Engenharia Química, UFBA, Salvador-BA, Fone: (71) 99978- 3702,francisleisantos@yahoo.com.br
Apresentado no
Congresso Técnico Científico da Engenharia e da Agronomia – CONTECC’2016
29 de agosto a 1 de setembro de 2016 – Foz do Iguaçu, Brasil
RESUMO: As tecnologias convencionais de obtenção do óxido de grafeno (OG) são de baixo rendimento e baixa reprodutibilidade. O baixo rendimento aumenta o preço do produto final. O grama do OG chega a ser vendido por $ 150 no mercado internacional. O alto preço do OG restringe as pesquisas aplicadas com esse material a poucos laboratórios especializados em nanotecnologia. O alto preço do óxido de grafeno e as limitações tecnológicas atuais inibem sua aplicação em larga escala pelas indústrias nacionais e estrangeiras. Objetiva-se por este trabalho, apresentar um novo processo de produção de óxido de grafeno (OG) a temperatura ambiente. O novo processo em patente é caracterizado pela oxirredução de uma substância rica em carbono a temperatura ambiente. A nova rota de processo pode ser usada para obtenção do OG nas escalas de laboratório e industrial. O produto obtido pela nova rota de processo de produção de OG foi caracterizado por Espectroscopia Raman, MEV, DRX e Microscopia Óptica. Os resultados mostram a formação de compostos do tipo grafíticos (característicos de OG) e carbono amorfo (aC).
PALAVRAS–CHAVE: óxido de grafeno, métodos de produção, carbono amorfo.
NEW PROCESS FOR PRODUCTION OF GRAPHENE OXIDE AT ROOM TEMPERATURE
ABSTRACT: Conventional technologies for obtaining graphene oxide (OG) are low-income and low reproducibility. The low yield increases the price of the final product. Gram’s OG comes to be sold for $ 150 on the international market. The high price of OG restricts the applied research with this material a few specialized laboratories in nanotechnology. The high price of the graphene oxide and the current technology limitations inhibit its large-scale application of national industry and foreign. The objective for this work is to present a new graphene oxide production process (GL) at room temperature. The new process is characterized by the patent redox carbon-rich material at room temperature. The new process route can be used for obtaining GL on laboratory and industrial scale. The product obtained by the new route OG production process was characterized by Raman spectroscopy, SEM, XRD and optical microscopy. Results show the formation of graphitic compounds of the type (characteristic OG) and amorphous carbon (aC). KEYWORDS: graphene oxide, production methods, amorphous carbon.
INTRODUÇÃO
Atualmente, tem-se utilizado o termo grafeno de forma um pouco mais ampla, abrangendo não só o material original (formado por uma única folha com espessura monoatômica), mas também a uma família de materiais formados por duas, três, quatro folhas de grafeno empilhadas de forma organizada cujas propriedades são diferentes entre si, devido às diferentes interações entre as várias folhas em
cada estrutura (Mehl, 2014). O óxido de grafeno reduzido ou simplesmente grafeno, como é mais conhecido atualmente, foi o primeiro cristal bidimensional estável isolado, com seu longo sistema pconjugado, onde os elétrons estão confinados em duas dimensões, o que confere propriedades excepcionais (Zarbine et all., 2013) tais como condutividade elétrica, resistência mecânica, leveza entre outras peculiaridades que diversificam a sua aplicação.
O grafeno é o alótropo mais novo da família do carbono ao lado do diamante e do carbono amorfo. O grafeno foi isolado e caracterizado pela primeira vez em 2004, através de sucessivas etapas de PEELING de um pedaço de grafite, com o auxílio de uma fita adesiva. O trabalho rendeu o prêmio Nobel de física de 2010 a seus autores, A. Geim e K. Novoselov, da Universidade de Manchester.
(Zarbine et all, 2013).Outro método muito difundido de obtenção do grafeno a partir do grafite se chama ESFOLIAÇÂO QUÌMICA, onde procura enfraquecer as ligações que unem as folhas de grafeno.Existem mais de uma rota de processo estudada por vários pesquisadores, porém a mais conhecida usa o grafite natural como substância rica em carbono. Esse grafite é aquecido em solução ácida, (H2SO4, HCl entre outros) e ainda utilizando-se do recurso de micro-ondas para produzir o óxido de grafite. Este óxido, já com as folhas de grafeno distanciadas umas das outras, é lavado com água deionizada e submetido a um processo de esfoliação (separação das folhas) utilizando-se
ultrassom ou a expansão térmica. Formando assim, o óxido de grafeno que pode ser reduzido com hidrazina para obtenção do grafeno ou óxido de grafeno reduzido. Neste método, o grafeno é submetido a um processo agressivo que acaba provocando vários defeitos na estrutura molecular. Os defeitos inviabilizam a produção do grafeno em larga escala além de diminuir a qualidade desejada
através da ESFOLIAÇÂO QUÌMICA.
As rotas apresentadas acima são de baixo rendimento e alto impacto ambiental pela extração do grafite natural obtido pelo processo de extração mineral. Outra forma de obter o grafite que é através do processo CVD (Carbon Vapour Deposition). O CVD é um método que sintetiza o grafite a partir de gases ricos em carbono, em especial o etileno que é aquecido a 1400 oC em um forno especial. Nesta etapa, o grafeno se forma em filme único sobre um substrato metálico (cobre), porém ainda não se conseguiu produzir grafeno em larga escala com áreas maiores através do método CVD. Fato este que limita a reprodutibilidade do método em questão. Poucos países, por exemplo: EUA, China e Cingapura, produzem o óxido de grafeno em larga escala e comercializam a altos preços que variam de $50 a $250/ grama a depender da pureza. Esta realidade restringe as pesquisas aplicadas com esse material a poucos laboratórios no mundo e inibem os investimentos em inovação; apesar do comprovado potencial de impacto nos setores de energia, biomedicina, agricultura, industrial,
eletrônicos, energias renováveis, ambiental, aeronáutico, aplicações médicas, têxteis. A importância desse material é proporcional ao alto número de patentes publicadas nos últimos anos. Zarbin et all.(2013) afirma que o principal desafio ainda está no desenvolvimento de métodos massivos de produção de amostras de óxido de grafeno reduzido com qualidade estrutural, e com controle do número de camadas. Afirma Mehl (2014) que há um grande interesse no desenvolvimento de rotas de preparação do grafeno, que sejam viáveis do ponto de vista prático (com alto rendimento e pureza, com e boa qualidade estrutural) e economicamente viável.
Esse trabalho foi resultado do estudo individual sobre eletrólise, nanotecnologia e interação molecular que deram as bases teóricas para a construção do protótipo em funcionamento. O novo método brasileiro, cujo título da patente é “PRODUção de nanopartículas de carbono a temperatura ambiente” (BR 10 2016012475 1) foi denominado de “ROTA NHK” que neste trabalho foi usado para
obtenção de OG. O destaque do novo método está na reprodutibilidade, na baixa emissão de poluentes e controle das etapas de produção. O objetivo desse trabalho é apresentar um novo processo de produção do óxido de grafeno a temperatura ambiente.
MATERIAL E MÉTODOS
A substância rica em carbono usada neste novo processo de produção de óxido de grafeno foi o carbono natural; e constitui o eletrólito de uma célula eletrolítica onde será submetido aos processos de oxidação e redução.Os testes experimentais foram realizados em instalação adaptada a um laboratório que está, atualmente, localizado na cidade de Alagoinhas-BA. As pesquisas foram financiadas com recursos próprios.
O produto obtido pela “ROTA NHK”, foi analisado por espectroscopia Raman empregando-se um espectrômetro Raman Witec (Alpha 500), acoplado a um detector CCD Witec (modelo DV401ABV-352) do Laboratório de Corrosão e Proteção do IPT. A focalização do laser na amostra e a coleta da radiação espalhada foram feitas através de um microscópio óptico Witec (Carl Zeiss, Serien-Nr
334000409). Foi utilizada linha de excitação na região do infravermelho próximo em 785,0 nm de um laser de diodo (XTRA 00222, Toptica) e na região do visível em 532,0 nm de um laser de argônio (WiTec) e em 633,0 nm de um laser de hélio-neônio (Modelo 30584, Optics Inc.). Os espectros são resultado da média de dez espectros obtidos com tempo de integração de 1 s, utilizando-se rede de
difração de 600 linhas/mm; lente de aumento de 100x (número de abertura 0,55, CF Plan). A Figura 1 a seguir representa o novo método de obtenção de óxido de grafeno a temperatura ambiente. Onde mostra uma fonte rica em carbono natural (1), na concentração de 0,576g/ml que constitui o meio reacional. A carga positiva constitui o anodo (3) de prata onde ocorrerá a etapa de
oxidação das partículas de carbono para obtenção do grafito oxidado em suspensão. Em paralelo, o outro eletrodo inerte (ex: Ag, Au) é alimentado com carga negativa e constitui o catodo (3) onde ocorrerá a reação de redução do grafito oxidado a óxido de grafeno reduzido em suspensão. Cada eletrodo é conectado por fios condutores de eletricidade (5) e são imersos no reator (2) de 30ml de capacidade com massa total de 15g aproximadamente. Uma fonte de energia elétrica (4) gera a diferença de potencial (ddp) necessário para o processo de oxirredução. O tempo de reação estimado para esse processo é de 9,0 min/ml de suspensão na CNTP em recipiente fechado, podendo variar em função da concentração. O óxido de grafeno quando exposto à luz excessiva sofre degradação. A Figura 1 representa o sistema da nova rota do processo em narrativa.
Figura 1 – ROTA NHK de produção de óxido de grafeno a temperatura ambiente.
RESULTADOS E DISCUSSÃO
O novo processo de produção de óxido de grafeno é caracterizado pela reação redox na mistura reacional que origina a formação de nano partículas de óxido de grafeno em suspensão no reator que opera a temperatura ambiente. Toda a carga de carbono é convertida em alótropos. As vantagens são a redução do custo energético, redução na geração de resíduos, controle das etapas de produção e reprodutibilidade do processo. A Figura 2 mostra o produto final em suspensão aquosa (A) cuja concentração mássica do produto obtido foi de aproximadamente de 0,600 g/ml em suspensão, as imagens (B) e (C) são o MEV e o DRX do produto obtido pelo novo método.
Figura 2 – (A) Nanopartículas de OG em suspensão aquosa, (B) MEV e (C) DRX da amostra.
A respeito dos testes para caracterização por espectroscopia Raman da amostra de carbono verificou que amostra apresenta elevada sensibilidade às radiações empregadas, o que resulta em sua degradação durante a irradiação com laser, mesmo em potência mínima (Figura 3) indicando que a amostra é fotossensível e pode formar outros alótropos do carbono a exemplo do carbono amorfo. Figura 3 – Imagens de microscopia óptica em aumento de 100x da Amostra de OG antes e após irradiação por laser para obtenção do espectro Raman nos comprimentos de onda (A) 532,0 nm, (B) 633,0 nm e (C) 785,0 nm.
Devido à sensibilidade da amostra, espectros Raman foram adquiridos com tempo curto de aquisição (50 ms) e 10 acumulações de maneira a diminuir o tempo de exposição à radiação do laser. Desta maneira, a qualidade dos espectros, razão sinal ruído, é baixa (Figura 4). O padrão espectral na página seguinte, para uma mesma radiação excitante, varia de acordo com o ponto de análise, indicando que a amostra não é homogênea (Figuras 4B e 4C) podendo conter óxido de grafeno e outros alótropos do carbono. Os espectros apresentados nas Figuras 4A e 4B apresentam duas bandas largas em aproximadamente 1350 cm-1 e 1580 cm-1 . Estas bandas podem ser atribuídas às bandas G e D de compostos sp2 de carbono como o óxido de grafeno, respectivamente (Dresselhaus et all. , 2010). Estas bandas também são observadas nos espectros de carbono amorfo (Marton et all., 2013). Para os espectros nas radiações 532,0 nm e 633,0 nm (Figura 4A e 4B) a banda D (~1380 cm-1 ) é mais intensa que a banda G (~1350 cm-1 ). Esta razão de intensidade está de acordo com aquela observada para compostos grafíticos, tais como óxidos de grafeno (Stankovich et all., 2007). A elevada largura das bandas G e D indica a presença de carbono amorfo. Os compostos sp2 de carbono (grafite, OG, grafeno, nanotubos de carbono, fulerenos) apresentam efeito Raman ressonante. No efeito Raman ressonante, a energia do fóton da radiação excitante é comparável ou mesmo coincidente com a energia de transição eletrônica da molécula em estudo (cromóforo). Neste caso, ocorre uma intensificação na ordem de 105 vezes das bandas associadas aos modos vibracionais do cromóforo. Por conta disso, não é possível afirmar, a partir dos espectros Raman, qual a proporção entre o óxido de grafeno e o carbono amorfo presentes na amostra analisada.
A amostra apresenta elevada sensibilidade ao laser empregado para caracterização por espectroscopia Raman. Os espectros Raman nas radiações excitante 532,0 nm e 633,0 nm apresentam bandas largas em aproximadamente 350 cm-1 e 1580 cm-1 , sendo a segunda mais intensa que a primeira. Tais resultados indicam que a amostra contem compostos do tipo grafítico (característico do óxido de grafeno) e carbono amorfo, porém não permite determinar se as estruturas grafíticas de óxido de grafeno estão presentes em elevada concentração na amostra devido ao efeito Raman ressonante. Este fato mostra a oportunidade de melhoria nas próximas etapas do nosso trabalho.
CONCLUSÕES
O novo método produz óxido de grafeno à temperatura ambiente.
AGRADECIMENTOS
-Deus pelos desafios que a vida impôs.
-Minha Família pelo apoio e confiança depositados neste trabalho.
-IF/UFBA e ao DEQ/UFCG pelas caracterizações do MEV e DRX respectivamente.
REFERÊNCIAS
Dresselhaus, M.S.; Jorio, A.; Hofmann, M.; Dresselhaus, G.; Saito, R. Perspectives on carbono
nanotubes and graphene Raman Spectroscopy. Nano letters, v. 10, p. 751-758, 2010.
Li, X.; Cai, W.; An, J.; Kim, S.; Nah, J.; Yong, D.; Piner, R.; Veldmakanni, A.; Juerg, I.; Tutuk, E.;
Banerjee, S. K.; Colombo, L.; Ruoff, R. S.; Science, p.324 e p.1312, 2009.
Marton, M.; Vojs, M.; Zdravecká, E.; Himmerlich, M.; Haensel, T.; Krishock, S.; Michiniak, P.;
Vesely, M.; Redhammer, R. Raman spectroscopy of amorphous carbono prepared by pulsed arc
discharge in various gas mixture. Journal of Spectroscopy, v. 2013, 6p. 2013.
Mehl, Hiany. The effect of variation of reactional parameters in the preparation of graphene by
oxidation and reduction of graphite. Quím. Nova, vol.37, n.10, pp.1639-1645. 2014.
Stankovich, S.; Dikin, D.A.; Piner, R.D; Kohlhaas, K.A.; Kleinhammes, A.; Jia, Y.; Wu, Y.; Nguyen,
S.T.; Rouff, R.S. Synthesis of graphene-based nanosheets via chemical reduction of exfoliated
grafite oxide. Carbon, v. 45, p. 1558-1565, 2007.
Zarbin, Aldo J. G. e Oliveira, Marcela M.. Carbon nanostructures (nanotubes and graphene): Quo
Vadis?. Quím. Nova, vol.36, n.10, pp.1533-1539. 2013.
ALLEN ATLAS CEREBRAL
Visão geral
Esta base de dados de células cerebrais é uma pesquisa de características biológicas derivadas de dados de células únicas, de humanos e ratos.
O banco de dados contém propriedades eletrofisiológicas , morfológicas e transcriptômicas coletadas de células individuais e modelos simulando atividade celular. Nesta fase inicial da geração de dados, a cobertura da pesquisa foi focada em áreas selecionadas do córtex cerebral e nos neurônios talâmicos.
Procure os dados de resposta eletrofisiológica e as morfologias neuronais reconstruídas usando a ferramenta de Pesquisa de Caracteres Celulares . Os dados transcriptômicos podem ser acessados através da página de download .
Use o Kit de Desenvolvimento de Software Allen (SDK) para acessar e analisar programaticamente dados em bruto e executar modelos.
Os dados podem ser baixados selecionando experimentos individuais na ferramenta de Pesquisa de Caracteres de Celular, acessando arquivos RNA-Seq transcriptômicos através da página de Download , ou através do SDK ou API Allen
Dados de rato
As células são adquiridas de áreas cerebrais selecionadas em ratos adultos. As células são identificadas para o isolamento com base em linhas de ratos transgênicos que abrigam repórteres fluorescentes conduzidos por drivers específicos do tipo celular. Para análises eletrofisiológicas e morfológicas, foram selecionadas células excitatórias com expressão enriquecida em camada e células inibitórias baseadas em marcadores clássicos. As áreas cerebrais selecionadas para análise incluem sub-regiões do córtex visual, córtex motor e córtex motor lateral anterior (ALM), na área do motor secundário (MOs).
Para a análise transcriptômica, foram realizadas dissecções regionais e laminares em espécimes de linhas transgênicas pan-neuronais, pan-excitatórias e pan-inibitórias, para amostra de forma abrangente. Os dados do núcleo geniculado lateral (LGd) também estão incluídos.
Este diagrama interativo de Venn mostra quantas células estão disponíveis para cada modalidade de dados (eletrofisiologia, morfologia, transcriptômica) e modelos. Selecione uma categoria para visualizar o subconjunto de células.
Existem 1058 células de mouse para as quais temos dados de eletrofisiologia.
Dados humanos
As células são adquiridas a partir do tecido cerebral doado nos lobos temporais ou frontais com base em anotações estruturais descritas no Atlas de referência do cérebro humano Allen . Para análises eletrofisiológicas e morfológicas no córtex, as células são selecionadas com base na forma do soma e na localização laminar.
Para a análise transcriptômica, diferentes camadas de córtex são dissecadas e os núcleos neuronais são isolados. A amostragem laminar é guiada pelo número relativo de neurônios presentes em cada camada.
Sobre eletrofisiologia
As gravações de grampos de patch de células inteiras fornecem informações básicas sobre propriedades de disparo celular. As gravações são realizadas usando uma variedade de protocolos de estímulo, incluindo pulsos curtos, passos longos, rampas lentas e ruído naturalista para caracterizar as propriedades intrínsecas desses neurônios. Os protocolos detalhados são descritos no whitepaper técnico de visão geral de eletrofisiologia .
Sobre Morfologia
A estrutura celular informa a função e a diversidade neuronal. Para ver a forma da célula, as células são preenchidas com biocitina e imagens em série para visualizar suas morfologias. Imagens planar e reconstruções de células 3D podem ser visualizadas com os dados de eletrofisiologia da célula, ou baixados para análise off-line. Os protocolos detalhados são descritos no whitepaper técnico da síntese de morfologia .
Sobre Transcriptomics
A seqüência de ARN pode fornecer um perfil transcriptômico para cada célula. Os transcritos genéticos são isolados, amplificados e seqüenciados, e as leituras estão alinhadas com um genoma de referência. A expressão de ARN por gene é relatada como uma média de isoformas de transcrição. Os dados estão disponíveis para células inteiras e, em alguns casos, isolados de frações nucleares. Para os núcleos, uma proporção significativa de lições se alinha aos intrões. Todos os dados podem ser baixados e os protocolos detalhados são descritos no quadro técnico geral da transcriptomics .
Sobre Modelos
Uma variedade de modelos neuronais que simulam propriedades de células intrínsecas estão disponíveis. Os modelos incluem: modelos generalizados de injeção e fogo, modelos biofisicamente realistas, de neurônio único com dendritos passivos e soma ativo (perisomático) e com condutâncias ativas (tudo ativo). As simulações podem ser vistas em linha ao lado das respostas celulares medidas, quando disponíveis. Todos os modelos podem ser baixados, e protocolos detalhados são descritos nos whitepapers técnicos: GLIF , perisomatic , all-active .

DNALinux – Uma solução Linux para Bioinformática
Programas de bioinformática incluídos no DNALinux
- ApE- A Plasmid Editor
- AutodockSuite 4.0.1
- Biopython 1.50
- Blast 2.2.20
- ClustalX
- Emboss 6.0.1
- EMNU-1.05
- ESIM4-1.0.0
- FinchTV
- HMMER-2.3.2
- IPRSCAN-4.3.1
- Kalign 2.03
- MEMENEW-0.1.0
- MIRA-2.8.2
- MSE-1.0.0
- MYEMBOSS-6.0.0
- NCBI Toolkit
- PHYLIP NEW-3.67
- Polyxmass 0.9.7
- primer3 1.1.4 / primer3plus
- Pymol 1.0r2
- Rasmol 2.7.3.1
- Readseq 1.7
- Sigma Align 1.1.1
- ASSINATURA-0.1.0
- ESTRUTURA-0.1.0
- TREEFINDER
- TreeView 0.5.1
- TOPO-1.0.0
- Vienna RNA 1.8.25
O ApE- A Plasmid Editor
Funciona no Windows (testado em 98, XP, NT), OS X (10.3, 10.4 e 10.5) e Linux / Unix, destaca sites de restrição na janela de edição, mostra tradução, Tm,% GC, ORF de DNA selecionado em tempo real, lê os arquivos DNA Strider, Fasta, Genbank e EMBL, copiar e salvar gráficos como metarquivos do Windows (somente MS Windows) em outros recursos. Para mais informações, visite o website.
Web site: http://www.biology.utah.edu/jorgensen/wayned/ape/
Outros documentos:
Citação:
AutodockSuite 4.0.1
AutoDock é um conjunto de ferramentas de encaixe automatizadas. Ele é projetado para prever como pequenas moléculas, como substratos ou medicamentos candidatos, se ligam a um receptor de estrutura 3D conhecida. O AutoDock consiste de dois programas principais: AutoDock executa o encaixe do ligando para um conjunto de grades descrevendo a proteína alvo; O AutoGrid pré-calcula essas grades. Além de usá-los para encaixe, as redes de afinidade atômica podem ser visualizadas. Isso pode ajudar, por exemplo, a orientar químicas sintéticas orgânicas para designar melhores aglutinantes.
Web site: http://autodock.scripps.edu/
Outra documentação:
Citação: Morris GM, Goodsell DS, Huey R, Olson AJ. Distribuição automated docking de ligandos flexíveis para proteínas: aplicações paralelas do AutoDock 2.4. J Comput Aided Mol Des. 1996 Ago; 10 (4): 293-304. Pubmed
O Biopython é um conjunto de ferramentas livremente disponíveis para computação biológica escrita em Python por uma equipe internacional de desenvolvedores.
É um esforço colaborativo distribuído para o desenvolvimento de bibliotecas e aplicações Python que abordam as necessidades dos trabalhos atuais e futuros em bioinformática. O código fonte está disponível sob a licença Biopython, que é extremamente liberal e compatível com quase todas as licenças do mundo. Trabalhamos junto com a Open Bioinformatics Foundation, que generosamente fornece espaço na web e CVS para o projeto.
Web site: www.biopython.org
Outra documentação: Bassi S (2007) A Primer on Python for Life Science Researchers. PLoS Comput Biol 3 (11): e199. doi: 10.1371 / journal.pcbi.0030199
Python para Bioinformáticalivro de Sebastian Bassi
Citação: Cock PJ, Antao T, Chang JT, Chapman BA, Cox CJ, Dalke A, Friedberg I, Hamelryck T, Kauff F, Wilczynski B e Hoon MJ. Biopython: ferramentas Python livremente disponíveis para biologia molecular computacional e bioinformática. Bioinformática 2009 1 de junho; 25 (11) 1422-3. doi: 10.1093 / bioinformática / btp163 pmid: 19304878. Pubmed
Blast 2.2.20
A Ferramenta de Pesquisa de Alinhamento Local Básico (BLAST) encontra regiões de similaridade local entre sequências. O programa compara sequências de nucleotídeos ou proteínas para bases de dados de sequências e calcula a significância estatística dos fósforos. O BLAST pode ser usado para inferir relações funcionais e evolutivas entre sequências, bem como ajudar a identificar membros de famílias de genes.
Site: http://blast.ncbi.nlm.nih.gov/
Outros documentos:
Citação: Altschul, Stephen F., Thomas L. Madden, Alejandro A. Schaffer, Jinghui Zhang, Zheng Zhang, Webb Miller e David J Lipman (1997), “Gapped BLAST e PSI-BLAST: uma nova geração de programas de pesquisa de banco de dados de proteína”, Nucleic Acids Res. 25: 3389-3402.
ClustalX
Alinhamento múltiplo de sequências de ácido nucleico e proteína.
Clustal X é uma interface do Windows para o programa de alinhamento de sequências múltiplas ClustalW. Ele fornece um ambiente integrado para realizar alinhamentos múltiplos de seqüências e perfis e analisar os resultados. O alinhamento da seqüência é exibido em uma janela na tela. Um esquema de coloração versátil foi incorporado, permitindo destacar os recursos conservados no alinhamento. Os menus pull-down na parte superior da janela permitem selecionar todas as opções necessárias para o alinhamento de múltiplas sequências e perfis tradicionais.
Site: http://www.clustal.org/
Outros documentos:
Citação: Jeanmougin, F., Thompson, JD, Gouy, M., Higgins, DG e Gibson, TJ (1998) Alinhamento de seqüências múltiplas com Clustal X. Tendências Biochem Sci, 23, 403-5.
O EMBOSS é “A Suite Européia de Software de Biologia Molecular Européia”. O EMBOSS é um pacote gratuito de análise de software de código aberto especialmente desenvolvido para as necessidades da comunidade de usuários de biologia molecular (por exemplo, EMBnet). O software lida automaticamente com dados em uma variedade de formatos e até permite a recuperação transparente de dados de sequência da web. Além disso, à medida que as extensas bibliotecas são fornecidas com o pacote, é uma plataforma para permitir que outros cientistas desenvolvam e liberem softwares com um verdadeiro espírito de código aberto. O EMBOSS também integra uma série de pacotes e ferramentas atualmente disponíveis para a análise de seqüências em um todo sem costura. O EMBOSS quebra a tendência histórica para pacotes de software comercial.
Web site: www.emboss.org
Outra documentação:
Citação: Arroz, P. Longden, eu. e Bleasby, A. EMBOSS: The European Molecular Biology Open Software Suite (2000) Tendências em Genética 16, (6) pp276-277.
EMNU-1.05
emnu exibe um menu simples baseado em caracteres que permite que você exiba os nomes dos programas em relevo e selecione-os. Ele também exibe os nomes dos arquivos em seu diretório atual e permite exibir seus conteúdos, copiá-los, excluí-los e fazer outras coisas com eles. Emnu permite mover os menus de programas ou arquivos usando as teclas de seta. Pressionando RETURN quando você selecionou um item executará um programa ou exibirá um arquivo.
Web site: http://web.mit.edu/emboss_v4.0.0/www/embassy/emnu/emnu.html
Outros documentos:
Citação:
ESIM4-1.0.0
Alinhar um mRNA a um
site de sequência de DNA genômico : http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/esim4/esim4.html
Outros documentos:
Citação: Florea L, Hartzell G, Zhang Z, Rubin GM, Miller W. “Um programa de computador para alinhar uma sequência de cDNA com uma sequência de DNA genômico”. Genoma Res 1998 Sep; 8 (9): 967-74
FinchTV – Visualizador de rastreamento de cromatografia de sequência de DNA.
O utilitário FinchTV para visualização de arquivos de cromatograma é um aplicativo autônomo que os pesquisadores usam para visualizar e editar facilmente seus dados de seqüência com interatividade dinâmica.
Web site: http://www.geospiza.com/Products/finchtv.shtml
Outros documentos:
Citação:
HMMER-2.3.2 Os
modelos escondidos do Markov do perfil (HMMs do perfil) podem ser usados para fazer pesquisa de banco de dados sensível usando descrições estatísticas do consenso de uma família de seqüência. HMMER é uma implementação livremente distribuível do software HMM de perfil para análise de seqüência de proteínas.
Web site: http://hmmer.janelia.org/
Outra documentação:
Citação: A teoria por trás do perfil HMMs: R. Durbin, S. Eddy, A. Krogh e G. Mitchison, análise de seqüência biológica: modelos probabilísticos de proteínas e ácidos nucleicos, Cambridge University Press, 1998. IPRSCAN-4.3.1 Web site: documentação adicional: Citação:
Kalign 2.03
Kalign é uma ferramenta de linha de comando para realizar o alinhamento múltiplo de seqüências biológicas. Ele emprega o algoritmo Wu-Manber string-matching, para melhorar tanto a precisão quanto a velocidade do alinhamento. Ele usa abordagem de alinhamento global e progressivo, enriquecendo empregando um algoritmo aproximado de correspondência de cadeias para calcular distâncias de seqüência e incorporando combinações locais no alinhamento de outra forma global. Nas comparações feitas por seus autores, Kalign era cerca de 10 vezes mais rápido do que o ClustalW e, dependendo do tamanho do alinhamento, até 50 vezes mais rápido do que os métodos iterativos populares.
Web site: http://msa.sbc.su.se/cgi-bin/msa.cgi
Outra documentação:
Citação: Timo Lassmann e Erik LL Sonnhammer. Kalign – um algoritmo de alinhamento de sequências múltiplo preciso e rápido. BMC Bioinformatics 2005, 6: 298doi: 10.1186 / 1471-2105-6-298
MEMENEW-0.1.0 Web site de
detecção de motivos
: http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/memenew/ememe.html
MIRA-2.8.2
O assembler do fragmento mira genome é um montador especializado para projetos de seqüenciamento classificados como “difíceis” devido ao alto número de repetições similares. Para transcrições de EST, miraEST é especializada na reconstrução de transcritos de ARNm prístinos enquanto detecta e classifica polimorfismos de nucleotídeos únicos (SNP) ocorrendo em diferentes variações.
O sistema de montagem está usando estratégias multipassos iterativas centradas no uso de regiões de alta confiança dentro de seqüências e tem uma estratégia de retorno para usar regiões de baixa confiança quando necessário.
Site: http://chevreux.org/projects_mira.html
MSE-1.0.0 Web site do
Editor de Sequências Múltiplas
: http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/mse/mse.html
MYEMBOSS-6.0.0 O
MYEMBOSS fornece uma estrutura de diretório e stubs de makefile para desenvolver suas próprias aplicações mais facilmente do que em versões anteriores a 3.0.0.
Web site: http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/myemboss/
NCBI Toolkit
O NCBI Toolkit é uma coleção de utilitários desenvolvidos para a produção e distribuição do GenBank, Entrez, BLAST e serviços relacionados pelo Centro Nacional de Informação Biotecnológica. Inclui as ferramentas populares de bioinformática formatdb e blastall.
Web site: http://www.ncbi.nih.gov/IEB/ToolBox/SDKDOCS/INDEX.HTML
Polyxmass 0.9.7
PHYLIP (o pacote de inferência PHYLogeny) é um pacote de programas para inferir filogenias (árvores evolutivas). Está disponível gratuitamente na Internet e escrito para trabalhar em tantos tipos diferentes de sistemas de computador quanto possível.
Felsenstein, J. 2005. PHYLIP (Phylogeny Inference Package) versão 3.6. Distribuído pelo autor. Departamento de Ciências do Genoma, Universidade de Washington, Seattle.
Web site: http://evolution.gs.washington.edu/phylip.html
polyxmass é um conjunto de software para espectrometria de massa (bio) de polímero que é o Software Livre desenvolvido no GNU / Linux. Permite a definição de química de polímeros e a simulação / análise de dados espectrométricos de massa obtidos em (bio) polímeros.
Citação: Filippo Rusconi, GNU polyxmass: uma estrutura de software para simulações de espectrometria de massa de analitos lineares (bio-) poliméricos. BMC Bioinformatics 2006, 7: 226doi: 10.1186 / 1471-2105-7-226
Primer3 1.1.4 / Primer3plus (GUI da Web para primer3) O
Primer3 é um programa amplamente utilizado para projetar iniciadores de PCR (PCR = “Reação em Cadeia de Polimerase”). O PCR é uma ferramenta essencial e omnipresente em genética e biologia molecular. O Primer3 também pode projetar sondas de hibridação e iniciadores de seqüenciamento.
Web site: http://primer3.sourceforge.net/
Outra documentação:
Citação: Rozen S, Skaletsky H (2000) Primer3 na WWW para usuários em geral e para programadores biologicos. Em: Krawetz S, Misener S (eds) Métodos e Protocolos de Bioinformática: Métodos em Biologia Molecular. Humana Press, Totowa, NJ, pp 365-386
Pymol 1.0r2 O
PyMOL é um sistema de visualização molecular de código aberto, criado por Warren Lyford DeLano e comercializado pela DeLano Scientific LLC, que é uma empresa de software privada dedicada à criação de ferramentas úteis que se tornam universalmente acessíveis para comunidades científicas e educacionais.
Site: http://www.pymol.org/
Rasmol 2.7.3.1
RasMol é um programa de gráficos moleculares destinado à visualização de proteínas, ácidos nucleicos e moléculas pequenas.
Site: http://www.rasmol.org/
readseq 1.7
Lê e grava seções nucleicas / proteínas em vários formatos. Os arquivos de dados podem ter múltiplas seqüências.
Sigma Align 1.1.1
A maioria das ferramentas para o alinhamento de sequências múltiplas são focadas no alinhamento da seqüência de proteínas ou da sequência de DNA que codifica a proteína. Sigma (“Alinhamento múltiplo codicioso simples”) é um programa de alinhamento com um novo algoritmo e esquema de pontuação projetado especificamente para seqüência de DNA não codificante.
Site: http://www.imsc.res.in/~rsidd/sigma/
Outros documentos:
Citação: Rahul Siddharthan, “Sigma: alinhamento múltiplo de sequências de DNA não codificantes fracamente conservadas”, BMC Bioinformatics 7: 143 ( 2006). Pubmed
SIGNATURE-0.1.0
Web site: http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/signature/
STRUCTURE-0.1.0
Web site: http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/structure/
TREEFINDER calcula árvores filogenéticas a partir de seqüências moleculares. O programa infere árvores mesmo grandes por máxima probabilidade sob uma variedade de modelos de evolução de seqüência. Ele aceita dados de nucleotídeos e aminoácidos e leva em consideração a heterogeneidade da taxa. Modelos separados podem ser assumidos para partições de dados definidas pelo usuário, taxas separadas, comprimentos de borda separados, composições de caracteres separadas. Todos os parâmetros podem ser estimados a partir dos dados. A pesquisa de árvores pode ser guiada por restrições topológicas fornecidas pelo usuário e iniciar árvores.
Web site: http://www.treefinder.de/
Outros documentos: o manual está disponível em formato pdf .
Citação: Jobb, G., A. von Haeseler e K. Strimmer. TREEFINDER: Um poderoso ambiente de análise gráfica para filogenética molecular. 2004. BMC Evolutionary Biology. Pubmed
TreeView 0.5.1
TreeView fornece uma maneira simples de ver o conteúdo de um arquivo de árvore de formato NEXUS, PHYLIP, Hennig86, Clustal ou outro formato. Enquanto PAUP e MacClade possuem excelentes instalações de impressão de árvores, pode haver momentos em que você só deseja ver as árvores sem ter que carregar o conjunto de dados de que foram geradas. O pacote PHYLIP contém programas de desenho de árvores que oferecem uma maior variedade de árvores que o TreeView, mas são um tanto desgastantes de usar. O próximo PAUP * para Windows não possui uma interface gráfica, portanto, o TreeView permite que você crie árvores de qualidade de publicação a partir de arquivos PAUP, diretamente ou gerando arquivos gráficos para edição por outros programas.
Web site: http://taxonomy.zoology.gla.ac.uk/rod/treeview.html
Outra documentação:
Citação: Page, RDM 1996. TREEVIEW: uma aplicação para exibir árvores filogenéticas em computadores pessoais. Aplicações informáticas nas Biociências 12: 357-358.
TOPO-1.0.0
TOPO cria uma imagem de uma proteína transmembranar.
Web site: http://saf.bio.caltech.edu/hhmi_manuals/solaris/embassy_apps/topo/topo.html
Vienna RNA 1.8.25
O Vienna RNA Package consiste em uma biblioteca de código C e vários programas autônomos para a predição e comparação de estruturas secundárias de RNA.
A predição da estrutura secundária do RNA através da minimização de energia é a função mais utilizada na embalagem. Nós fornecemos três tipos de algoritmos de programação dinâmica para a predição da estrutura: o algoritmo mínimo de energia livre de (Zuker & Stiegler 1981) que produz uma única estrutura ótima, o algoritmo de função de partição de (McCaskill 1990) que calcula as probabilidades de pares de bases no conjunto termodinâmico, e o algoritmo de dobragem sub-óptimo de (Wuchty et.al 1999) que gera todas as estruturas sub-ótimas dentro de uma determinada faixa de energia da energia ideal. Para a comparação de estrutura secundária, o pacote contém várias medidas de distância (dissimilaridades) usando o alinhamento de cordas ou a edição de árvores (Shapiro & Zhang, 1990). Finalmente, fornecemos um algoritmo para projetar seqüências com uma estrutura predefinida (dobra inversa).
Web site: http://www.tbi.univie.ac.at/RNA/
Outros documentos:
Citação: Ivo L. Hofacker, Walter Fontana, Peter F. Stadler, L. Sebastian Bonhoeffer, Manfred Tacker e Peter Schuster. Folding Rápido e Comparação de Estruturas Secundárias de RNA. Apareceu em: Monatsh.Chem. 125: 167-188 (1994).
Faça o download do DNALinux
DNALinux Virtual Desktop. Python for Bioinformatics Edition (NOVO! Jun 2009)
 DNALinux VDE (arquivo Torrent)
DNALinux VDE (arquivo Torrent)
Instruções de instalação
- Baixe o arquivo torrent .
- Faça o download do DNALinux usando o torrent com um programa compatível com bittorrent como “Bittorrent”, Bittorrnado, Vuze ( veja aqui para obter uma lista completa )
- Faça o download do VMWare Free Player
- Baixar 7zip uncompressor
- Descompacte DNALinux.7z usando o 7zip e reproduza o arquivo vmk com o VMWare Player.
Download direto no RapidShare (12 partes):
Instrução para arquivos baixados do Rapidshare:
Para juntar as peças no Windows:
copiar / b dbasea * dnalinux.7z
Para juntar as peças no Linux:
gato dbasea *> dnalinux.7z
Soma de verificação MD5: f75e88f48e08161be62b70a8ef465e17
Versão antiga (não suportada):
Servidor DNALinux
Nota: A nova versão (VDE Python para Bioinformatcs) já é um servidor, portanto a edição do servidor é interrompida.